readr, tibble e tidyr
Um modelo de ciência de dados
Uma forma esquemática, mas útil de encarar o processo de análise de dados e de produção de conhecimento a partir de informações secundárias é esta figura:
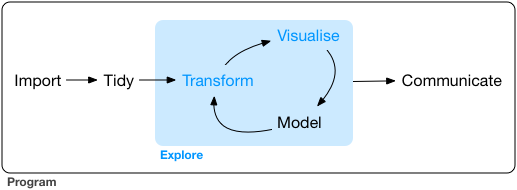
Ela acaba sendo um ponto de partida interessante porque contextualiza a maioria dos pacotes que vamos utilizar. O readr é uma maneira de facilitar e tornar mais rápida a importação de dados em formatos comuns, como o texto delimitado por separadores e o formato colunado com larguras-fixas utilizado pelo IBGE.
A tibble é uma proposta de modernização para o data.frame, aproveitando a estrutura flexível e poderosa, mas mudando certas convenções, como a conversão de strings para fatores, permitindo a criação de colunas de listas, utilização de nomes mais complexos para colunas, etc.
O tidyr é uma forma de reformatar (reshape) bancos de dados que vêm em formatos que dificultam seu processamento, permitindo que o analista facilmente reconfigure a informação para o formato mais adequado. Ele é o primeiro pacote que veremos sobre a parte de “cozinha” dos dados, no qual é preciso processar o dado bruto para torná-lo passível de análise.
O mesmo pode ser dito para os outros pacotes que veremos nos próximos dias. Cada um deles foi pensado para resolver um problema ou facilitar uma rotina de trabalho relacionada com um dos passos descritos acima.
Programação literária
Vários dos conceitos que guiam o design do tidyverse são orientados por uma certa filosofia. Esta filosofia dita que a principal preocupação por trás de um programa não é que ele funcione. Eventualmente e com um pouco de persistência, é quase sempre possível chegar a uma solução técnica adequada. A principal questão é que o programa é uma ferramenta de comunicação com outras pessoas que o lerão, seja seus colegas, colaboradores, alunos ou você mesmo, daqui a 2 anos, quando lembrar que você já escreveu um programa para aquela tarefa. Trago aqui alguns trechos de Literate Programming, de Donald Knuth (1984), traduzidos livremente por este que vos fala:
Creio que chegou a hora de melhorar significativamente a documentação dos programas, e que podemos atingir este objetivo tratando programas como obras literárias. Por isso o título: “Programação Literária”. Abandonemos os velhos hábitos de construir programas: ao invés de imaginar que nossa tarefa principal é instruir um computador sobre o que deve ser feito, concentremo-nos em explicar aos seres humanos o que queremos que o computador faça. O praticante da programação literária pode ser visto como um ensaísta, cuja principal preocupação é com a clareza da exposição e a excelência no estilo. Tal autor, com um dicionário na mão, escolhe os nomes das variáveis com cuidado e explica claramente seu significado. Ele ou ela esmera por um programa que é compreensível porque seus conceitos foram introduzidos na melhor ordem possível para o entendimento humano, utilizando uma mistura de métodos formais e informais que se complementam.
Assim, várias das escolhas feitas na construção de tidyverse procuram reforçar essa característica de interpretabilidade dos programas, como a ordem lógica das operações com o pipe (%>%), funções com nomes mais longos e semânticos, a utilização de uma função específica para cada tarefa, ao invés da adaptação de funções genéricas para uma grande variedade de tarefas. O propósito de um código escrito como tidyverse é que, no limite, a própria síntaxe do código funcione como parte da documentação do programa.
O mistério do pipe: %>%
Usuários de longa data do R já podem ter encontrado no mato esse animal estranho e podem ter ficado confusos com seu significado. O pipe é de origem humilde e nasceu nos sistemas Unix há muitas décadas atrás. Seu objetivo é muito simples: e se você tiver uma sequência de computações em que cada uma recebe o resultado daquela feita anteriormente? Claro que você poderia fazer:
x <- 1:10
y <- diff(x)
mean(y)
## [1] 1
Mas não seria interessante pular os objetos intermediários e ir direto ao ponto? O pipe vem do pacote magrittr, mas ele vêm carregado em quase todos os pacotes do tidyverse. Vamos carregar logo o tibble.
# Para ter acesso ao pipe, basta carregar um pacote do tidyverse, como tibble, dplyr, tidyr, etc.
# Vamos carregar o tibble agora
library(tibble)
x %>% diff() %>% mean()
## [1] 1
Vejamos um exemplo mais real, quantos artistas existem no dataset billboard? Podemos usar unique e length para descobrir.
x <- unique(tidyr::billboard$artist)
length(x)
## [1] 228
Mas com %>% fica bem melhor:
tidyr::billboard %>% .$artist %>% unique() %>% length()
## [1] 228
Ok, mas como ele funciona? É simples, o pipe carrega o objeto a sua esquerda num ponto . invisível que é automaticamente passado como o primeiro argumento da função à direita.
x <- 1:10
mean(x)
## [1] 5.5
x %>% mean()
## [1] 5.5
OK, mas e se meu argumento não for o primeiro, ainda posso usar pipe? Pode! É só usar explicitamente um ponto no lugar onde você quer aproveitar o efeito:
iris %>% boxplot(Sepal.Length ~ Species, data = .)
O ponto . depois de data indica indica que ali deve ser colocado o iris. O pipe é uma peça chave de muitas funções do tidyverse, não porque ele seja obrigatório, mas sim porque ele permite expressar sequências de operações numa ordem mais lógica, do tipo: “Primeiro faça a, então b, então c, … “, ao contrário da forma como isto é geralmente feito usando parênteses para precedência.
# Compare
mean(diff(1:10))
## [1] 1
1:10 %>% diff %>% mean
## [1] 1
E assim fica desmistificado o mistério do pipe! Um último pulo do gato: pelo amor de deus ninguém digita Shift + %, >, Shift + %, basta usar o atalho: Ctrl + Shift + M que ele põe um pipe separado por espaços %>%.
readr
Usuários do R provavelmente vão estar familiarizados com os nossos leitores de arquivos mais comuns: read.table e read.csv. Talvez muitos de vocês já até memorizaram alguns dos argumentos mais comuns. Não é o caso aqui de revisitar esta função, mas o readr tem muitos paralelos com elas, porque é pensado como uma nova versão da mesma coisa.
# Comecemos carregando o readr
library(readr)
Debaixo do capô
O readr, como as funções de leitura do base é uma coleção de parsers, que transformam texto em objetos R com o tipo desejado.
parse_number(c("1", "20", "38"))
## [1] 1 20 38
parse_character(c("banana", "maçã", "pêra"))
## [1] "banana" "ma<e7><e3>" "p<ea>ra"
# Note os acentos e caracteres especiais
parse_character(c("banana", "maçã", "pêra"),
locale = locale(encoding = "Windows-1252"))
## [1] "banana" "maçã" "pêra"
parse_logical(c("true", "false", "true"))
## [1] TRUE FALSE TRUE
Em geral, a gente não precisa descer tanto o nível, a gente vai trabalhar mesmo é com os leitores de dados “retangulares”. Como os do base, eles são read_csv, read_table, etc. Vamos trabalhar com bancos de dados que já vêm no pacote, para facilitar o processo.
# Lista os datasets que vem no pacote
readr_example()
## [1] "challenge.csv" "epa78.txt" "example.log"
## [4] "fwf-sample.txt" "massey-rating.txt" "mtcars.csv"
## [7] "mtcars.csv.bz2" "mtcars.csv.zip"
Uma coisa que gosto de fazer é olhar como o arquivo está organizado antes de tentar abrí-lo. Muitos de vocês podem fazer isso com readLines. Ela ganhou sua versão no pacote com read_lines.
# Vamos tentar abrir massey-rating.txt
read_lines(readr_example("massey-rating.txt"), n_max = 10)
## [1] "UCC PAY LAZ KPK RT COF BIH DII ENG ACU Rank Team Conf"
## [2] " 1 1 1 1 1 1 1 1 1 1 1 Ohio St B10 "
## [3] " 2 2 2 2 2 2 2 2 4 2 2 Oregon P12 "
## [4] " 3 4 3 4 3 4 3 4 2 3 3 Alabama SEC "
## [5] " 4 3 4 3 4 3 5 3 3 4 4 TCU B12 "
## [6] " 6 6 6 5 5 7 6 5 6 11 5 Michigan St B10 "
## [7] " 7 7 7 6 7 6 11 8 7 8 6 Georgia SEC "
## [8] " 5 5 5 7 6 8 4 6 5 5 7 Florida St ACC "
## [9] " 8 8 9 9 10 5 7 7 10 7 8 Baylor B12 "
## [10] " 9 11 8 13 11 11 12 9 14 9 9 Georgia Tech ACC "
# Identificando o separador, escolho a função adequada
read_table(readr_example("massey-rating.txt"))
##
## -- Column specification --------------------------------------------------------
## cols(
## UCC = col_double(),
## PAY = col_double(),
## LAZ = col_double(),
## KPK = col_double(),
## RT = col_double(),
## COF = col_double(),
## BIH = col_double(),
## DII = col_double(),
## ENG = col_double(),
## ACU = col_double(),
## Rank = col_double(),
## Team = col_character(),
## Conf = col_character()
## )
## # A tibble: 10 x 13
## UCC PAY LAZ KPK RT COF BIH DII ENG ACU Rank Team Conf
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr> <chr>
## 1 1 1 1 1 1 1 1 1 1 1 1 Ohio~ B10
## 2 2 2 2 2 2 2 2 2 4 2 2 Oreg~ P12
## 3 3 4 3 4 3 4 3 4 2 3 3 Alab~ SEC
## 4 4 3 4 3 4 3 5 3 3 4 4 TCU B12
## 5 6 6 6 5 5 7 6 5 6 11 5 Mich~ B10
## 6 7 7 7 6 7 6 11 8 7 8 6 Geor~ SEC
## 7 5 5 5 7 6 8 4 6 5 5 7 Flor~ ACC
## 8 8 8 9 9 10 5 7 7 10 7 8 Bayl~ B12
## 9 9 11 8 13 11 11 12 9 14 9 9 Geor~ ACC
## 10 13 10 13 11 8 9 10 11 9 10 10 Miss~ SEC
Como identifiquei que as colunas estavam separadas por espaços, utilizei read_table cujo delimitador é o espaço " ".
A segunda feature mais interessante do readr, é uma interface para selecionar os tipos de colunas que serão importadas. Vejamos o seguinte exemplo.
# Vamos abrir mtcars.csv
read_lines(readr_example("mtcars.csv"), n_max = 10)
## [1] "\"mpg\",\"cyl\",\"disp\",\"hp\",\"drat\",\"wt\",\"qsec\",\"vs\",\"am\",\"gear\",\"carb\""
## [2] "21,6,160,110,3.9,2.62,16.46,0,1,4,4"
## [3] "21,6,160,110,3.9,2.875,17.02,0,1,4,4"
## [4] "22.8,4,108,93,3.85,2.32,18.61,1,1,4,1"
## [5] "21.4,6,258,110,3.08,3.215,19.44,1,0,3,1"
## [6] "18.7,8,360,175,3.15,3.44,17.02,0,0,3,2"
## [7] "18.1,6,225,105,2.76,3.46,20.22,1,0,3,1"
## [8] "14.3,8,360,245,3.21,3.57,15.84,0,0,3,4"
## [9] "24.4,4,146.7,62,3.69,3.19,20,1,0,4,2"
## [10] "22.8,4,140.8,95,3.92,3.15,22.9,1,0,4,2"
# Identificamos o separador de colunas, selecionamos a função adequada
read_csv(readr_example("mtcars.csv"))
##
## -- Column specification --------------------------------------------------------
## cols(
## mpg = col_double(),
## cyl = col_double(),
## disp = col_double(),
## hp = col_double(),
## drat = col_double(),
## wt = col_double(),
## qsec = col_double(),
## vs = col_double(),
## am = col_double(),
## gear = col_double(),
## carb = col_double()
## )
## # A tibble: 32 x 11
## mpg cyl disp hp drat wt qsec vs am gear carb
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 21 6 160 110 3.9 2.62 16.5 0 1 4 4
## 2 21 6 160 110 3.9 2.88 17.0 0 1 4 4
## 3 22.8 4 108 93 3.85 2.32 18.6 1 1 4 1
## 4 21.4 6 258 110 3.08 3.22 19.4 1 0 3 1
## 5 18.7 8 360 175 3.15 3.44 17.0 0 0 3 2
## 6 18.1 6 225 105 2.76 3.46 20.2 1 0 3 1
## 7 14.3 8 360 245 3.21 3.57 15.8 0 0 3 4
## 8 24.4 4 147. 62 3.69 3.19 20 1 0 4 2
## 9 22.8 4 141. 95 3.92 3.15 22.9 1 0 4 2
## 10 19.2 6 168. 123 3.92 3.44 18.3 1 0 4 4
## # ... with 22 more rows
O console nos mostra que a leitura do banco foi completada, mas também mostra Column Specification. Isto indica qual o tipo de dado que foi identificado automaticamente numa análise feita pela função guess_parser. Em diversos casos, nós podemos querer identificar manualmente as colunas. Vejamos um exemplo:
# Vamos identificar as colunas com spec
spec_csv(readr_example("mtcars.csv"))
##
## -- Column specification --------------------------------------------------------
## cols(
## mpg = col_double(),
## cyl = col_double(),
## disp = col_double(),
## hp = col_double(),
## drat = col_double(),
## wt = col_double(),
## qsec = col_double(),
## vs = col_double(),
## am = col_double(),
## gear = col_double(),
## carb = col_double()
## )
## cols(
## mpg = col_double(),
## cyl = col_double(),
## disp = col_double(),
## hp = col_double(),
## drat = col_double(),
## wt = col_double(),
## qsec = col_double(),
## vs = col_double(),
## am = col_double(),
## gear = col_double(),
## carb = col_double()
## )
# Copia e cola, modifica as colunas que queremos alterar
spec_cols <- cols(
mpg = col_double(),
cyl = col_factor(), # N de cilindros do automóvel
disp = col_double(),
hp = col_double(),
drat = col_double(),
wt = col_double(),
qsec = col_double(),
vs = col_double(),
am = col_factor(c("0", "1")), # Transmissão automática ou manual
gear = col_double(),
carb = col_double()
)
df <- read_csv(readr_example("mtcars.csv"), col_types = spec_cols)
df
## # A tibble: 32 x 11
## mpg cyl disp hp drat wt qsec vs am gear carb
## <dbl> <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <fct> <dbl> <dbl>
## 1 21 6 160 110 3.9 2.62 16.5 0 1 4 4
## 2 21 6 160 110 3.9 2.88 17.0 0 1 4 4
## 3 22.8 4 108 93 3.85 2.32 18.6 1 1 4 1
## 4 21.4 6 258 110 3.08 3.22 19.4 1 0 3 1
## 5 18.7 8 360 175 3.15 3.44 17.0 0 0 3 2
## 6 18.1 6 225 105 2.76 3.46 20.2 1 0 3 1
## 7 14.3 8 360 245 3.21 3.57 15.8 0 0 3 4
## 8 24.4 4 147. 62 3.69 3.19 20 1 0 4 2
## 9 22.8 4 141. 95 3.92 3.15 22.9 1 0 4 2
## 10 19.2 6 168. 123 3.92 3.44 18.3 1 0 4 4
## # ... with 22 more rows
# Para importar apenas colunas selecionadas, utilize 'cols_only()'
spec_cols2 <- cols_only(
mpg = col_double(),
cyl = col_factor(), # N de cilindros do automóvel
am = col_factor(c("0", "1")), # Transmissão automática ou manual
gear = col_double(),
carb = col_double()
)
df2 <- read_csv(readr_example("mtcars.csv"), col_types = spec_cols2)
df2
## # A tibble: 32 x 5
## mpg cyl am gear carb
## <dbl> <fct> <fct> <dbl> <dbl>
## 1 21 6 1 4 4
## 2 21 6 1 4 4
## 3 22.8 4 1 4 1
## 4 21.4 6 0 3 1
## 5 18.7 8 0 3 2
## 6 18.1 6 0 3 1
## 7 14.3 8 0 3 4
## 8 24.4 4 0 4 2
## 9 22.8 4 0 4 2
## 10 19.2 6 0 4 4
## # ... with 22 more rows
# Para indicar os tipos de colunas de um jeito mais sucinto, utilize uma string:
df <- read_csv(readr_example("mtcars.csv"), col_types = "dfddddddfdd")
# Só cuidado pra não perder a conta dos ds!
df
## # A tibble: 32 x 11
## mpg cyl disp hp drat wt qsec vs am gear carb
## <dbl> <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <fct> <dbl> <dbl>
## 1 21 6 160 110 3.9 2.62 16.5 0 1 4 4
## 2 21 6 160 110 3.9 2.88 17.0 0 1 4 4
## 3 22.8 4 108 93 3.85 2.32 18.6 1 1 4 1
## 4 21.4 6 258 110 3.08 3.22 19.4 1 0 3 1
## 5 18.7 8 360 175 3.15 3.44 17.0 0 0 3 2
## 6 18.1 6 225 105 2.76 3.46 20.2 1 0 3 1
## 7 14.3 8 360 245 3.21 3.57 15.8 0 0 3 4
## 8 24.4 4 147. 62 3.69 3.19 20 1 0 4 2
## 9 22.8 4 141. 95 3.92 3.15 22.9 1 0 4 2
## 10 19.2 6 168. 123 3.92 3.44 18.3 1 0 4 4
## # ... with 22 more rows
Você também pode querer definir características de localização, como a codificação de caracteres, os separadores de decimal e de milhar e etc. A melhor forma de fazer isso é definir um locale.
meu_locale <- locale(encoding = "UTF-8", decimal_mark = ",", grouping_mark = ".")
Aí é só passar isso pra uma das funções do pacote sob o argumento locale
read_csv(readr_example("mtcars.csv"), locale = meu_locale)
##
## -- Column specification --------------------------------------------------------
## cols(
## mpg = col_number(),
## cyl = col_double(),
## disp = col_number(),
## hp = col_double(),
## drat = col_number(),
## wt = col_number(),
## qsec = col_number(),
## vs = col_double(),
## am = col_double(),
## gear = col_double(),
## carb = col_double()
## )
## # A tibble: 32 x 11
## mpg cyl disp hp drat wt qsec vs am gear carb
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 21 6 160 110 39 262 1646 0 1 4 4
## 2 21 6 160 110 39 2875 1702 0 1 4 4
## 3 228 4 108 93 385 232 1861 1 1 4 1
## 4 214 6 258 110 308 3215 1944 1 0 3 1
## 5 187 8 360 175 315 344 1702 0 0 3 2
## 6 181 6 225 105 276 346 2022 1 0 3 1
## 7 143 8 360 245 321 357 1584 0 0 3 4
## 8 244 4 1467 62 369 319 20 1 0 4 2
## 9 228 4 1408 95 392 315 229 1 0 4 2
## 10 192 6 1676 123 392 344 183 1 0 4 4
## # ... with 22 more rows
Existe ainda a possibilidade de ler dados colunados com largura-fixa. readr implementa quatro funções diferentes para ajudar na construção do dicionário:
# Nossos dados
x <- readr_example("fwf-sample.txt")
read_lines(x, n_max = 10)
## [1] "John Smith WA 418-Y11-4111"
## [2] "Mary Hartford CA 319-Z19-4341"
## [3] "Evan Nolan IL 219-532-c301"
# separados por espaço
dic1 <- fwf_empty(x)
dic1
## $begin
## [1] 0 5 20 30
##
## $end
## [1] 4 13 22 NA
##
## $skip
## [1] 0
##
## $col_names
## [1] "X1" "X2" "X3" "X4"
df <- read_fwf(file = x, col_positions = dic1)
##
## -- Column specification --------------------------------------------------------
## cols(
## X1 = col_character(),
## X2 = col_character(),
## X3 = col_character(),
## X4 = col_character()
## )
df
## # A tibble: 3 x 4
## X1 X2 X3 X4
## <chr> <chr> <chr> <chr>
## 1 John Smith WA 418-Y11-4111
## 2 Mary Hartford CA 319-Z19-4341
## 3 Evan Nolan IL 219-532-c301
# indicando a largura da coluna
larguras <- c(20, 10, 12)
dic2 <- fwf_widths(larguras)
dic2
## # A tibble: 3 x 3
## begin end col_names
## <dbl> <dbl> <chr>
## 1 0 20 X1
## 2 20 30 X2
## 3 30 42 X3
df <- read_fwf(file = x, col_positions = dic2)
##
## -- Column specification --------------------------------------------------------
## cols(
## X1 = col_character(),
## X2 = col_character(),
## X3 = col_character()
## )
df
## # A tibble: 3 x 3
## X1 X2 X3
## <chr> <chr> <chr>
## 1 John Smith WA 418-Y11-4111
## 2 Mary Hartford CA 319-Z19-4341
## 3 Evan Nolan IL 219-532-c301
# indicando onde cada coluna começa e termina
comeca <- c(1, 21, 30)
termina <- c(20, 29, 42)
dic3 <- fwf_positions(comeca, termina)
dic3
## # A tibble: 3 x 3
## begin end col_names
## <dbl> <dbl> <chr>
## 1 0 20 X1
## 2 20 29 X2
## 3 29 42 X3
df <- read_fwf(file = x, col_positions = dic3)
##
## -- Column specification --------------------------------------------------------
## cols(
## X1 = col_character(),
## X2 = col_character(),
## X3 = col_character()
## )
df
## # A tibble: 3 x 3
## X1 X2 X3
## <chr> <chr> <chr>
## 1 John Smith WA 418-Y11-4111
## 2 Mary Hartford CA 319-Z19-4341
## 3 Evan Nolan IL 219-532-c301
# indicando pares nome-valor
dic4 <- fwf_cols(
nome = c(1, 20),
uf = c(21, 29),
numero = c(30, 42))
dic4
## # A tibble: 3 x 3
## begin end col_names
## <int> <int> <chr>
## 1 0 20 nome
## 2 20 29 uf
## 3 29 42 numero
df <- read_fwf(file = x, col_positions = dic4)
##
## -- Column specification --------------------------------------------------------
## cols(
## nome = col_character(),
## uf = col_character(),
## numero = col_character()
## )
df
## # A tibble: 3 x 3
## nome uf numero
## <chr> <chr> <chr>
## 1 John Smith WA 418-Y11-4111
## 2 Mary Hartford CA 319-Z19-4341
## 3 Evan Nolan IL 219-532-c301
Especificar dicionários para arquivos colunados é um pé-no-saco, por sorte, existem pacotes que já fizeram parte desse trabalho por nós. O readr não melhora muita o serviço manual de construção de dicionários, o que ele oferece é um ganho de performance tremendo. read_fwf é centenas de vezes mais rápido que o base read.fwf.
Em termos do que o pacote faz, é basicamente isso. A única coisa que falta mencionar é que ele importa os dados como tibbles ao invés do data.frame padrão, mas isso já é um ótimo gancho pra nossa próxima parte.
tibbles
Tibbles são basicamente data.frames com um método mais bonitinho de print. Elas automaticamente se ajustam a largura da sua tela, omitindo as colunas que estouram, e por padrão imprimem só as 10 primeiras observações. Outras características que pessoalmente gosto, é que elas informam o tipo de variável junto com o nome, arrendondam digitos significativos, destacam números grandes, negativos, NAs e etc.
df <- read_csv(readr_example("mtcars.csv"), col_types = cols()) # omitir a especificação
df
## # A tibble: 32 x 11
## mpg cyl disp hp drat wt qsec vs am gear carb
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 21 6 160 110 3.9 2.62 16.5 0 1 4 4
## 2 21 6 160 110 3.9 2.88 17.0 0 1 4 4
## 3 22.8 4 108 93 3.85 2.32 18.6 1 1 4 1
## 4 21.4 6 258 110 3.08 3.22 19.4 1 0 3 1
## 5 18.7 8 360 175 3.15 3.44 17.0 0 0 3 2
## 6 18.1 6 225 105 2.76 3.46 20.2 1 0 3 1
## 7 14.3 8 360 245 3.21 3.57 15.8 0 0 3 4
## 8 24.4 4 147. 62 3.69 3.19 20 1 0 4 2
## 9 22.8 4 141. 95 3.92 3.15 22.9 1 0 4 2
## 10 19.2 6 168. 123 3.92 3.44 18.3 1 0 4 4
## # ... with 22 more rows
Do ponto de vista prático, elas funcionam exatamente igual a data.frames, tudo que você pode fazer com um data.frame, você faz com tibbles. O que elas trazem de novidade é:
- Elas nunca mudam o tipo de dado inputado.
df1 <- data.frame(x = list(1:5, 1:10, 1:20))
df1
## x.1.5 x.1.10 x.1.20
## 1 1 1 1
## 2 2 2 2
## 3 3 3 3
## 4 4 4 4
## 5 5 5 5
## 6 1 6 6
## 7 2 7 7
## 8 3 8 8
## 9 4 9 9
## 10 5 10 10
## 11 1 1 11
## 12 2 2 12
## 13 3 3 13
## 14 4 4 14
## 15 5 5 15
## 16 1 6 16
## 17 2 7 17
## 18 3 8 18
## 19 4 9 19
## 20 5 10 20
df2 <- tibble(x = list(1:5, 1:10, 1:20))
df2
## # A tibble: 3 x 1
## x
## <list>
## 1 <int [5]>
## 2 <int [10]>
## 3 <int [20]>
- Elas nunca ajustam os nomes das variáveis
names(data.frame(`nome hipster` = 1))
## [1] "nome.hipster"
names(tibble(`nome hipster` = 1))
## [1] "nome hipster"
- Ela avalia cada argumento de forma “preguiçosa” e sequencial
# erro
data.frame(x = 1:5, y = x ^ 2)
## Error in x^2: argumento não-numérico para operador binário
# funciona
tibble(x = 1:5, y = x ^ 2)
## # A tibble: 5 x 2
## x y
## <int> <dbl>
## 1 1 1
## 2 2 4
## 3 3 9
## 4 4 16
## 5 5 25
- Ela nunca utiliza row.names
head(data.frame(state.x77))
## Population Income Illiteracy Life.Exp Murder HS.Grad Frost Area
## Alabama 3615 3624 2.1 69.05 15.1 41.3 20 50708
## Alaska 365 6315 1.5 69.31 11.3 66.7 152 566432
## Arizona 2212 4530 1.8 70.55 7.8 58.1 15 113417
## Arkansas 2110 3378 1.9 70.66 10.1 39.9 65 51945
## California 21198 5114 1.1 71.71 10.3 62.6 20 156361
## Colorado 2541 4884 0.7 72.06 6.8 63.9 166 103766
as_tibble(state.x77)
## # A tibble: 50 x 8
## Population Income Illiteracy `Life Exp` Murder `HS Grad` Frost Area
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 3615 3624 2.1 69.0 15.1 41.3 20 50708
## 2 365 6315 1.5 69.3 11.3 66.7 152 566432
## 3 2212 4530 1.8 70.6 7.8 58.1 15 113417
## 4 2110 3378 1.9 70.7 10.1 39.9 65 51945
## 5 21198 5114 1.1 71.7 10.3 62.6 20 156361
## 6 2541 4884 0.7 72.1 6.8 63.9 166 103766
## 7 3100 5348 1.1 72.5 3.1 56 139 4862
## 8 579 4809 0.9 70.1 6.2 54.6 103 1982
## 9 8277 4815 1.3 70.7 10.7 52.6 11 54090
## 10 4931 4091 2 68.5 13.9 40.6 60 58073
## # ... with 40 more rows
- Ela muda a “regra da reciclagem”: apenas são aceitos vetores unitários ou vetores de tamanho igual aos demais
data.frame(x = 1:10, y = 1:5)
## x y
## 1 1 1
## 2 2 2
## 3 3 3
## 4 4 4
## 5 5 5
## 6 6 1
## 7 7 2
## 8 8 3
## 9 9 4
## 10 10 5
# erro
tibble(x = 1:10, y = 1:5)
## Error: Tibble columns must have compatible sizes.
## * Size 10: Existing data.
## * Size 5: Column `y`.
## i Only values of size one are recycled.
tibble(x = 1:10, y = 1)
## # A tibble: 10 x 2
## x y
## <int> <dbl>
## 1 1 1
## 2 2 1
## 3 3 1
## 4 4 1
## 5 5 1
## 6 6 1
## 7 7 1
## 8 8 1
## 9 9 1
## 10 10 1
tibble(x = 1:10, y = c(1:5, 1:5))
## # A tibble: 10 x 2
## x y
## <int> <int>
## 1 1 1
## 2 2 2
## 3 3 3
## 4 4 4
## 5 5 5
## 6 6 1
## 7 7 2
## 8 8 3
## 9 9 4
## 10 10 5
- Tibbles são estritas com relação as operações de subsetting com
[
df1 <- data.frame(x = 1:3, y = 3:1)
class(df1[,1:2])
## [1] "data.frame"
class(df1[,1])
## [1] "integer"
df2 <- tibble(x = 1:3, y = 3:1)
class(df2[, 1:2])
## [1] "tbl_df" "tbl" "data.frame"
class(df2[, 1])
## [1] "tbl_df" "tbl" "data.frame"
# Se quiser extrair só uma coluna, utilize '[[' ou '$'
class(df2[[1]])
## [1] "integer"
class(df2$x)
## [1] "integer"
Elas também não aceitam ‘partial matching’ de nomes de variáveis.
df <- data.frame(nome_de_cavalo = 1)
df$nome
## [1] 1
df2 <- tibble(nome_de_cavalo = 1)
df2$nome
## Warning: Unknown or uninitialised column: `nome`.
## NULL
tidyr
Ok, nossos dados estão no R, mas, muitas vezes, não estão no formato adequado. De maneira geral, analistas de dados vão dar preferência a um formato parecido com este:

Isto tem uma razão de ser que deve tornar-se óbvia quando tentarmos realizar as operações de transformação de variáveis, visualização, modelos, etc. Porém, muitas vezes outras considerações são feitas na hora registrar os dados, armazená-los, apresentá-los ao público, por isso, frequentemente nossos dados não estão no formato tidy e precisam ser reformatados. Essa é uma das principais tarefas do tidyr e é nela que vamos nos concentrar.
Atente que reformatação, como muitos outros aspectos da análise de dados, não é receita de bolo. Muitas vezes o formato desejado não é óbvio, muito menos os passos necessários para chegar lá. Porém, vou apresentar as ferramentas e alguns exemplos simples que cobrem muitos dos nossos casos de uso.
library(tidyr)
who
## # A tibble: 7,240 x 60
## country iso2 iso3 year new_sp_m014 new_sp_m1524 new_sp_m2534 new_sp_m3544
## <chr> <chr> <chr> <int> <int> <int> <int> <int>
## 1 Afghani~ AF AFG 1980 NA NA NA NA
## 2 Afghani~ AF AFG 1981 NA NA NA NA
## 3 Afghani~ AF AFG 1982 NA NA NA NA
## 4 Afghani~ AF AFG 1983 NA NA NA NA
## 5 Afghani~ AF AFG 1984 NA NA NA NA
## 6 Afghani~ AF AFG 1985 NA NA NA NA
## 7 Afghani~ AF AFG 1986 NA NA NA NA
## 8 Afghani~ AF AFG 1987 NA NA NA NA
## 9 Afghani~ AF AFG 1988 NA NA NA NA
## 10 Afghani~ AF AFG 1989 NA NA NA NA
## # ... with 7,230 more rows, and 52 more variables: new_sp_m4554 <int>,
## # new_sp_m5564 <int>, new_sp_m65 <int>, new_sp_f014 <int>,
## # new_sp_f1524 <int>, new_sp_f2534 <int>, new_sp_f3544 <int>,
## # new_sp_f4554 <int>, new_sp_f5564 <int>, new_sp_f65 <int>,
## # new_sn_m014 <int>, new_sn_m1524 <int>, new_sn_m2534 <int>,
## # new_sn_m3544 <int>, new_sn_m4554 <int>, new_sn_m5564 <int>,
## # new_sn_m65 <int>, new_sn_f014 <int>, new_sn_f1524 <int>,
## # new_sn_f2534 <int>, new_sn_f3544 <int>, new_sn_f4554 <int>,
## # new_sn_f5564 <int>, new_sn_f65 <int>, new_ep_m014 <int>,
## # new_ep_m1524 <int>, new_ep_m2534 <int>, new_ep_m3544 <int>,
## # new_ep_m4554 <int>, new_ep_m5564 <int>, new_ep_m65 <int>,
## # new_ep_f014 <int>, new_ep_f1524 <int>, new_ep_f2534 <int>,
## # new_ep_f3544 <int>, new_ep_f4554 <int>, new_ep_f5564 <int>,
## # new_ep_f65 <int>, newrel_m014 <int>, newrel_m1524 <int>,
## # newrel_m2534 <int>, newrel_m3544 <int>, newrel_m4554 <int>,
## # newrel_m5564 <int>, newrel_m65 <int>, newrel_f014 <int>,
## # newrel_f1524 <int>, newrel_f2534 <int>, newrel_f3544 <int>,
## # newrel_f4554 <int>, newrel_f5564 <int>, newrel_f65 <int>
Esse é um banco de dados difícil de analisar, ele tem 60 colunas, indicando o número de casos de tuberculose em diversos estágios da doença, por país e ano. O problema é que ao invés de termos algo como:
tribble(
~pais, ~ano, ~tipo, ~idade, ~casos,
"brasil", 1980, "extrapulmonar", "15-24", 10,
"brasil", 1990, "relapso", "15-24", 10
)
## # A tibble: 2 x 5
## pais ano tipo idade casos
## <chr> <dbl> <chr> <chr> <dbl>
## 1 brasil 1980 extrapulmonar 15-24 10
## 2 brasil 1990 relapso 15-24 10
As informações de tipo de tuberculose e idade dos pacientes estão espalhadas pelas colunas. Pra encurtar a história, precisamos “tombar” esse banco para que essas colunas se tornem um novo conjunto de variáveis. Vamos passo a passo.
# Primeiro, vamos excluir as colunas iso2 e iso3, porque elas são a mesma informação redundante
who$iso2 <- NULL
who$iso3 <- NULL
who1 <- pivot_longer(who,
cols = c(new_sp_m014:newrel_f65),
names_to = "tipo_tb",
values_to = "casos",
values_drop_na = TRUE)
who1
## # A tibble: 76,046 x 4
## country year tipo_tb casos
## <chr> <int> <chr> <int>
## 1 Afghanistan 1997 new_sp_m014 0
## 2 Afghanistan 1997 new_sp_m1524 10
## 3 Afghanistan 1997 new_sp_m2534 6
## 4 Afghanistan 1997 new_sp_m3544 3
## 5 Afghanistan 1997 new_sp_m4554 5
## 6 Afghanistan 1997 new_sp_m5564 2
## 7 Afghanistan 1997 new_sp_m65 0
## 8 Afghanistan 1997 new_sp_f014 5
## 9 Afghanistan 1997 new_sp_f1524 38
## 10 Afghanistan 1997 new_sp_f2534 36
## # ... with 76,036 more rows
Nosso primeiro passo é transformar todas as colunas de novos casos em um par de colunas:
colsindica quais colunas serão tombadas e quais serão mantidas.- Uma coluna
names_torecebe as categorias da variável. - Uma coluna
values_torecebe os valores das células. values_drop_naé uma opção para eliminar células vazias.
Essa primeira transformação já nos dá um banco de dados um pouco mais amigável, porém, ainda temos variáveis “presas” na coluna tipo_tb. Vamos tentar soltá-las.
# primeiro, corrigir uma pequena inconsistencia:
unique(who1$tipo_tb)
## [1] "new_sp_m014" "new_sp_m1524" "new_sp_m2534" "new_sp_m3544" "new_sp_m4554"
## [6] "new_sp_m5564" "new_sp_m65" "new_sp_f014" "new_sp_f1524" "new_sp_f2534"
## [11] "new_sp_f3544" "new_sp_f4554" "new_sp_f5564" "new_sp_f65" "new_sn_m014"
## [16] "new_sn_m1524" "new_sn_m2534" "new_sn_m3544" "new_sn_m4554" "new_sn_m5564"
## [21] "new_sn_m65" "new_ep_m014" "new_ep_m1524" "new_ep_m2534" "new_ep_m3544"
## [26] "new_ep_m4554" "new_ep_m5564" "new_ep_m65" "new_sn_f014" "newrel_m014"
## [31] "newrel_f014" "new_sn_f1524" "new_sn_f2534" "new_sn_f3544" "new_sn_f4554"
## [36] "new_sn_f5564" "new_sn_f65" "new_ep_f014" "new_ep_f1524" "new_ep_f2534"
## [41] "new_ep_f3544" "new_ep_f4554" "new_ep_f5564" "new_ep_f65" "newrel_m1524"
## [46] "newrel_m2534" "newrel_m3544" "newrel_m4554" "newrel_m5564" "newrel_m65"
## [51] "newrel_f1524" "newrel_f2534" "newrel_f3544" "newrel_f4554" "newrel_f5564"
## [56] "newrel_f65"
# Notem que newrel deveria ser new_rel
# Alguns de vocês devem conhecer 'gsub'
who1$tipo_tb <- gsub("newrel", "new_rel", who1$tipo_tb)
# Agora, podemos usar outra função chave do tidyr, 'separate'
who2 <- who1 %>% separate(col = tipo_tb,
into = c(NA, "tipo_tb", "sexo_idade"),
sep = "_")
who2
## # A tibble: 76,046 x 5
## country year tipo_tb sexo_idade casos
## <chr> <int> <chr> <chr> <int>
## 1 Afghanistan 1997 sp m014 0
## 2 Afghanistan 1997 sp m1524 10
## 3 Afghanistan 1997 sp m2534 6
## 4 Afghanistan 1997 sp m3544 3
## 5 Afghanistan 1997 sp m4554 5
## 6 Afghanistan 1997 sp m5564 2
## 7 Afghanistan 1997 sp m65 0
## 8 Afghanistan 1997 sp f014 5
## 9 Afghanistan 1997 sp f1524 38
## 10 Afghanistan 1997 sp f2534 36
## # ... with 76,036 more rows
# E outra passagem de separate para separar a idade do sexo
who3 <- who2 %>% separate(col = sexo_idade,
into = c("sexo", "idade"),
sep = 1)
who3
## # A tibble: 76,046 x 6
## country year tipo_tb sexo idade casos
## <chr> <int> <chr> <chr> <chr> <int>
## 1 Afghanistan 1997 sp m 014 0
## 2 Afghanistan 1997 sp m 1524 10
## 3 Afghanistan 1997 sp m 2534 6
## 4 Afghanistan 1997 sp m 3544 3
## 5 Afghanistan 1997 sp m 4554 5
## 6 Afghanistan 1997 sp m 5564 2
## 7 Afghanistan 1997 sp m 65 0
## 8 Afghanistan 1997 sp f 014 5
## 9 Afghanistan 1997 sp f 1524 38
## 10 Afghanistan 1997 sp f 2534 36
## # ... with 76,036 more rows
Bem melhor, não acham? Estamos agora com um banco de dados muito mais adequado para uma análise de dados em R. Cada linha é uma observação, cada coluna é uma informação sobre ela.
Alguns de vocês podem ter reparado que fizemos um caminho em que nosso banco de dados passou de ter muitas colunas para muitas linhas (ficou mais “longo”) e depois precisamos separar algumas das colunas que criamos em outras (o que fizemos com separate). Podemos facilmente imaginar situações em que queremos fazer o caminho inverso: transformar um banco do formato longo para o formato com mais colunas e unir colunas separadas em uma nova. Vamos ver um exemplo.
# Exemplo adaptado de https://en.wikipedia.org/wiki/List_of_countries_and_dependencies_by_population
populacao <- tribble(
~Rank, ~Country, ~Population, ~'% of world', ~Day, ~Month, ~Year, ~Source,
1L, "China", 1411778724, "17.9%", "1", "Nov", "2020", "Seventh Census on 2020",
2L, "India", 1377123716, "17.5%", "19", "May", "2021", "National population clock[3]",
3L, "United States", 331695937, "4.22%", "19", "May", "2021", "National population clock[4]",
4L, "Indonesia", 271350000, "3.45%", "31", "Dec", "2020", "National annual estimate[5]",
5L, "Pakistan", 225200000, "2.86%", "1", "Jul", "2021", "UN projection[2]",
6L, "Brazil", 213154869, "2.71%", "19", "May", "2021", "National population clock[6]",
7L, "Nigeria", 211401000, "2.69%", "1", "Jul", "2021", "UN projection[2]",
8L, "Bangladesh", 170689832, "2.17%", "19", "May", "2021", "National population clock[7]",
9L, "Russia", 146171015, "1.86%", "1", "Jan", "2021", "National annual estimate[8]",
10L, "Mexico", 126014024, "1.60%", "2", "Mar", "2020", "2020 census result[9]"
)
populacao
## # A tibble: 10 x 8
## Rank Country Population `% of world` Day Month Year Source
## <int> <chr> <dbl> <chr> <chr> <chr> <chr> <chr>
## 1 1 China 1411778724 17.9% 1 Nov 2020 Seventh Census o~
## 2 2 India 1377123716 17.5% 19 May 2021 National populat~
## 3 3 United Sta~ 331695937 4.22% 19 May 2021 National populat~
## 4 4 Indonesia 271350000 3.45% 31 Dec 2020 National annual ~
## 5 5 Pakistan 225200000 2.86% 1 Jul 2021 UN projection[2]
## 6 6 Brazil 213154869 2.71% 19 May 2021 National populat~
## 7 7 Nigeria 211401000 2.69% 1 Jul 2021 UN projection[2]
## 8 8 Bangladesh 170689832 2.17% 19 May 2021 National populat~
## 9 9 Russia 146171015 1.86% 1 Jan 2021 National annual ~
## 10 10 Mexico 126014024 1.60% 2 Mar 2020 2020 census resu~
Imagine que, por qualquer motivo, você prefira trabalhar com uma variável “Data” ao invés de dia, mês e ano. Podemos usar unite:
populacao2 <- populacao %>% unite(col = Data, Day, Month, Year, sep = " ")
populacao2
## # A tibble: 10 x 6
## Rank Country Population `% of world` Data Source
## <int> <chr> <dbl> <chr> <chr> <chr>
## 1 1 China 1411778724 17.9% 1 Nov 2020 Seventh Census on 2020
## 2 2 India 1377123716 17.5% 19 May 20~ National population cl~
## 3 3 United Stat~ 331695937 4.22% 19 May 20~ National population cl~
## 4 4 Indonesia 271350000 3.45% 31 Dec 20~ National annual estima~
## 5 5 Pakistan 225200000 2.86% 1 Jul 2021 UN projection[2]
## 6 6 Brazil 213154869 2.71% 19 May 20~ National population cl~
## 7 7 Nigeria 211401000 2.69% 1 Jul 2021 UN projection[2]
## 8 8 Bangladesh 170689832 2.17% 19 May 20~ National population cl~
## 9 9 Russia 146171015 1.86% 1 Jan 2021 National annual estima~
## 10 10 Mexico 126014024 1.60% 2 Mar 2020 2020 census result[9]
O outro problema que precisamos resolver frequentemente, é separar um par de variáveis em diversas colunas, fazendo o caminho inverso que fizemos no caso do dataset da OMS.
us_rent_income
## # A tibble: 104 x 5
## GEOID NAME variable estimate moe
## <chr> <chr> <chr> <dbl> <dbl>
## 1 01 Alabama income 24476 136
## 2 01 Alabama rent 747 3
## 3 02 Alaska income 32940 508
## 4 02 Alaska rent 1200 13
## 5 04 Arizona income 27517 148
## 6 04 Arizona rent 972 4
## 7 05 Arkansas income 23789 165
## 8 05 Arkansas rent 709 5
## 9 06 California income 29454 109
## 10 06 California rent 1358 3
## # ... with 94 more rows
No exemplo acima, queremos separar em colunas os valores das variáveis de renda e valor do aluguel. Esse tipo de operação tem um certo grau de abstração que me deu bastante dor de cabeça para entender inicialmente, então vamos olhar com carinho para o que queremos ter depois da transformação.
us_rent_income2 <- tribble(
~GEOID, ~NAME, ~income_estimate, ~rent_estimate, ~income_moe, ~rent_moe,
"01", "Alabama", 24476, 747, 136, 3,
"02", "Alaska", 32940, 1200, 508, 13
)
us_rent_income2
## # A tibble: 2 x 6
## GEOID NAME income_estimate rent_estimate income_moe rent_moe
## <chr> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 01 Alabama 24476 747 136 3
## 2 02 Alaska 32940 1200 508 13
O banco que queremos tem uma cara assim. Ele tem mais colunas novas e menos linhas, já que eu tinha no formato tidy uma variável chamada “variable” que guardava os valores renda e aluguel e duas colunas que guardavam os valores da estimativa e do moe. Agora, eu vou ter 4 colunas, duas para as estimativas de renda e aluguel e duas para os moes das mesmas variáveis. Como especificar isso para o banco todo? Usando pivot_wider.
us_rent_income %>% pivot_wider(names_from = variable, values_from = c(estimate, moe))
## # A tibble: 52 x 6
## GEOID NAME estimate_income estimate_rent moe_income moe_rent
## <chr> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 01 Alabama 24476 747 136 3
## 2 02 Alaska 32940 1200 508 13
## 3 04 Arizona 27517 972 148 4
## 4 05 Arkansas 23789 709 165 5
## 5 06 California 29454 1358 109 3
## 6 08 Colorado 32401 1125 109 5
## 7 09 Connecticut 35326 1123 195 5
## 8 10 Delaware 31560 1076 247 10
## 9 11 District of Columbia 43198 1424 681 17
## 10 12 Florida 25952 1077 70 3
## # ... with 42 more rows
As funções pivot_ tem diversos outros argumentos e cobrem diversos casos de uso. Vejam este exemplo da documentação de pivot_longer:
anscombe
## x1 x2 x3 x4 y1 y2 y3 y4
## 1 10 10 10 8 8.04 9.14 7.46 6.58
## 2 8 8 8 8 6.95 8.14 6.77 5.76
## 3 13 13 13 8 7.58 8.74 12.74 7.71
## 4 9 9 9 8 8.81 8.77 7.11 8.84
## 5 11 11 11 8 8.33 9.26 7.81 8.47
## 6 14 14 14 8 9.96 8.10 8.84 7.04
## 7 6 6 6 8 7.24 6.13 6.08 5.25
## 8 4 4 4 19 4.26 3.10 5.39 12.50
## 9 12 12 12 8 10.84 9.13 8.15 5.56
## 10 7 7 7 8 4.82 7.26 6.42 7.91
## 11 5 5 5 8 5.68 4.74 5.73 6.89
Podemos transformar esse banco de dados rapidamente usando um dos argumentos de pivot_longer, chamado names_pattern.
anscombe %>% pivot_longer(everything(),
names_to = c(".value", "set"),
names_pattern = "(.)(.)")
## # A tibble: 44 x 3
## set x y
## <chr> <dbl> <dbl>
## 1 1 10 8.04
## 2 2 10 9.14
## 3 3 10 7.46
## 4 4 8 6.58
## 5 1 8 6.95
## 6 2 8 8.14
## 7 3 8 6.77
## 8 4 8 5.76
## 9 1 13 7.58
## 10 2 13 8.74
## # ... with 34 more rows
Esse exemplo é interessante, porque ele se aproveita de uma “regular expression”, tema da parte do nosso curso em que falaremos sobre manipulação de strings com o stringr. Resumidas as contas, as colunas se chamam “x1, x2, x3 …” e a string “(.)(.)” indica que há dois “grupos” formados por um caractere cada. A string “.value” que vai no argumento de cima é um atalho da função para dizer “pegue o valor de todas as células das variáveis selecionadas”, aqui, todas. Ou seja, ele indica para a função que o primeiro caractere “x” ou “y” definirá uma nova variável e armazenará os valores das celulas, enquanto o segundo grupo “1”, “2”, “3” ou “4” formará uma segunda variável chamada “set” que contém apenas os nomes das colunas. Deu um nó na cabeça?
Uma última preocupação ao utilizar a reformatação de dados é o que ocorre com os valores NA. Vejamos este exemplo:
acoes <- tibble(
ano = c(2015, 2015, 2015, 2015, 2016, 2016, 2016),
qdr = c( 1, 2, 3, 4, 2, 3, 4),
lucro = c(1.88, 0.59, 0.35, NA, 0.92, 0.17, 2.66)
)
Existem dois tipos de valor nulo, explícito se diz de um valor nulo como aquele NA que aparece na variável lucro. Implícito é o valor que ocorre no primeiro quadrimestre de 2016, onde sequer foi adicionada uma linha no banco de dados. Os valores implícitos são muito sacanas, porque eles não são imediatamente visíveis.
acoes %>%
pivot_wider(names_from = ano, values_from = lucro)
## # A tibble: 4 x 3
## qdr `2015` `2016`
## <dbl> <dbl> <dbl>
## 1 1 1.88 NA
## 2 2 0.59 0.92
## 3 3 0.35 0.17
## 4 4 NA 2.66
Ao transformar o banco, o valor implícito ficou explícito. Caso você não esteja interessado neste valor, você pode passar o values_drop_na durante a transformação de volta ao formato original.
acoes %>%
pivot_wider(names_from = ano, values_from = lucro) %>%
pivot_longer(c(`2015`, `2016`),
names_to = "ano",
values_to = "lucro",
values_drop_na = TRUE)
## # A tibble: 6 x 3
## qdr ano lucro
## <dbl> <chr> <dbl>
## 1 1 2015 1.88
## 2 2 2015 0.59
## 3 2 2016 0.92
## 4 3 2015 0.35
## 5 3 2016 0.17
## 6 4 2016 2.66
Que faz os valores missing desaparecerem do resultado.
complete pode ser usada pra tornar valores implícitos, explícitos! A função toma todas as colunas pedidas e verifica todas as combinações possíveis de valores, preenchendo as lacunas com NA. Cuidado ao utilizar complete com valores numéricos ou conjuntos de colunas com muitas combinações possíveis, pois o número de combinações pode ser infinitamente grande e travar sua sessão!
acoes %>% complete(ano, qdr)
## # A tibble: 8 x 3
## ano qdr lucro
## <dbl> <dbl> <dbl>
## 1 2015 1 1.88
## 2 2015 2 0.59
## 3 2015 3 0.35
## 4 2015 4 NA
## 5 2016 1 NA
## 6 2016 2 0.92
## 7 2016 3 0.17
## 8 2016 4 2.66
Pra encerrar, fill serve para aqueles casos em que um valor missing indica que a última observação deve ser repetida. Pesquisadores brasileiros das antigas podem lembrar-se do Censo de 1991, em que o IBGE registrava os arquivos de domícilio e pessoas com esse sistema. Em inglês, isso se chama LOCF, ou “last observation carried forward”.
treatment <- tribble(
~ person, ~ treatment, ~response,
"Derrick Whitmore", 1, 7,
NA, 2, 10,
NA, 3, 9,
"Katherine Burke", 1, 4
)
treatment
## # A tibble: 4 x 3
## person treatment response
## <chr> <dbl> <dbl>
## 1 Derrick Whitmore 1 7
## 2 <NA> 2 10
## 3 <NA> 3 9
## 4 Katherine Burke 1 4
treatment %>% fill(person)
## # A tibble: 4 x 3
## person treatment response
## <chr> <dbl> <dbl>
## 1 Derrick Whitmore 1 7
## 2 Derrick Whitmore 2 10
## 3 Derrick Whitmore 3 9
## 4 Katherine Burke 1 4
tidyr tem também outras funcionalidades relevantes para modelagem estatística, mas acho que isso sai um pouco do escopo do curso. Quem sabe a gente não faz um curso posterior só sobre modelagem no tidyverse?
Revisão
readr
O pacote readr apresenta uma família de funções para substituir as funções do base relacionadas a importação de arquivos em formato texto, seja delimitado ou largura-fixa. São elas,
read_delimread_csvread_csv2read_tsvread_tableread_fwf
E assim sucessivamente. Durante o processo de importação, você pode querer especificar o tipo de coluna com cols ou cols_only, usando o argumento col_types. Ou use uma string do tipo “ddcdiDT” em que cada letra é um tipo de variável.
col_integercol_doublecol_factorcol_character
Etc. Você também pode querer definir características de localização, como a codificação de caracteres, os separadores de decimal e de milhar e etc. A melhor forma de fazer isso é definir um locale.
Ah, e você sempre pode salvar com write_, inclusive salvando/lendo compactado para bzip, gzip ou xzip.
tibble
Tibbles são uma versão do data.frame com algumas regrinhas novas. Vou apenas repetí-las aqui de forma resumida.
- tibbles tem um método print mais bonito e amigável, especialmente para bancos com muitas observações e variáveis.
- elas são estritas com operações de subsetting com
[e$. - elas não aceita a reciclagem de argumento de tamanho diferente de 1.
tidyr
tidyr é um pacote de reformatação de bancos, criando novas linhas e colunas a partir da reorganização das variáveis e valores existentes. Suas principais operações são:
pivot_longerpara converter colunas em linhaspivot_widerpara converter linhas em colunasseparatepara separar uma coluna em várias com base em caracteresunitepara unir diversas colunas em uma com base em caracteres
Ufa. Acabou né? Posso ir dormir já? Claro, só fazer uns exercícios!
Exercícios
- Como você importaria o banco “epa78.csv”
file <- readr_example("epa78.txt")
- Importe o banco “challenge.csv” e resolva os problemas com o tipo da coluna.
file <- readr_example("challenge.csv")
- Com o banco sala_aula dado a seguir, transforme-o para que ele contenha as variáveis nome, avaliação e nota.
sala_aula <- tribble(
~name, ~teste1, ~teste2, ~prova1,
"Billy", "<NA>", "D" , "C",
"Suzy", "F", "<NA>", "<NA>",
"Lionel", "B", "C" , "B",
"Jenny", "A", "A" , "B"
)
- Transforme o banco
relig_incomepara que ele contenha as colunas religião, renda e frequência.
relig_income
## # A tibble: 18 x 11
## religion `<$10k` `$10-20k` `$20-30k` `$30-40k` `$40-50k` `$50-75k` `$75-100k`
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Agnostic 27 34 60 81 76 137 122
## 2 Atheist 12 27 37 52 35 70 73
## 3 Buddhist 27 21 30 34 33 58 62
## 4 Catholic 418 617 732 670 638 1116 949
## 5 Don’t k~ 15 14 15 11 10 35 21
## 6 Evangel~ 575 869 1064 982 881 1486 949
## 7 Hindu 1 9 7 9 11 34 47
## 8 Histori~ 228 244 236 238 197 223 131
## 9 Jehovah~ 20 27 24 24 21 30 15
## 10 Jewish 19 19 25 25 30 95 69
## 11 Mainlin~ 289 495 619 655 651 1107 939
## 12 Mormon 29 40 48 51 56 112 85
## 13 Muslim 6 7 9 10 9 23 16
## 14 Orthodox 13 17 23 32 32 47 38
## 15 Other C~ 9 7 11 13 13 14 18
## 16 Other F~ 20 33 40 46 49 63 46
## 17 Other W~ 5 2 3 4 2 7 3
## 18 Unaffil~ 217 299 374 365 341 528 407
## # ... with 3 more variables: $100-150k <dbl>, >150k <dbl>,
## # Don't know/refused <dbl>
- Transforme o banco
billboardpara que ele contenha apenas uma coluna “semana” e uma coluna com a posição da música no ranking.
# Dica, você pode selecionar várias colunas usando o atalho wk1:wk76
billboard
## # A tibble: 317 x 79
## artist track date.entered wk1 wk2 wk3 wk4 wk5 wk6 wk7 wk8
## <chr> <chr> <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2 Pac Baby D~ 2000-02-26 87 82 72 77 87 94 99 NA
## 2 2Ge+her The Ha~ 2000-09-02 91 87 92 NA NA NA NA NA
## 3 3 Doors~ Krypto~ 2000-04-08 81 70 68 67 66 57 54 53
## 4 3 Doors~ Loser 2000-10-21 76 76 72 69 67 65 55 59
## 5 504 Boyz Wobble~ 2000-04-15 57 34 25 17 17 31 36 49
## 6 98^0 Give M~ 2000-08-19 51 39 34 26 26 19 2 2
## 7 A*Teens Dancin~ 2000-07-08 97 97 96 95 100 NA NA NA
## 8 Aaliyah I Don'~ 2000-01-29 84 62 51 41 38 35 35 38
## 9 Aaliyah Try Ag~ 2000-03-18 59 53 38 28 21 18 16 14
## 10 Adams, ~ Open M~ 2000-08-26 76 76 74 69 68 67 61 58
## # ... with 307 more rows, and 68 more variables: wk9 <dbl>, wk10 <dbl>,
## # wk11 <dbl>, wk12 <dbl>, wk13 <dbl>, wk14 <dbl>, wk15 <dbl>, wk16 <dbl>,
## # wk17 <dbl>, wk18 <dbl>, wk19 <dbl>, wk20 <dbl>, wk21 <dbl>, wk22 <dbl>,
## # wk23 <dbl>, wk24 <dbl>, wk25 <dbl>, wk26 <dbl>, wk27 <dbl>, wk28 <dbl>,
## # wk29 <dbl>, wk30 <dbl>, wk31 <dbl>, wk32 <dbl>, wk33 <dbl>, wk34 <dbl>,
## # wk35 <dbl>, wk36 <dbl>, wk37 <dbl>, wk38 <dbl>, wk39 <dbl>, wk40 <dbl>,
## # wk41 <dbl>, wk42 <dbl>, wk43 <dbl>, wk44 <dbl>, wk45 <dbl>, wk46 <dbl>,
## # wk47 <dbl>, wk48 <dbl>, wk49 <dbl>, wk50 <dbl>, wk51 <dbl>, wk52 <dbl>,
## # wk53 <dbl>, wk54 <dbl>, wk55 <dbl>, wk56 <dbl>, wk57 <dbl>, wk58 <dbl>,
## # wk59 <dbl>, wk60 <dbl>, wk61 <dbl>, wk62 <dbl>, wk63 <dbl>, wk64 <dbl>,
## # wk65 <dbl>, wk66 <lgl>, wk67 <lgl>, wk68 <lgl>, wk69 <lgl>, wk70 <lgl>,
## # wk71 <lgl>, wk72 <lgl>, wk73 <lgl>, wk74 <lgl>, wk75 <lgl>, wk76 <lgl>
-
Experimente fazer o caminho inverso dos exercícios 3 a 5, devolvendo os datasets ao seu formato original. O que você observou?
-
O que os argumentos
extraefillem separate fazem? Utilize o exemplo a seguir como guia.
tibble(x = c("a,b,c", "d,e,f,g", "h,i,j")) %>%
separate(x, c("um", "dois", "tres"))
## Warning: Expected 3 pieces. Additional pieces discarded in 1 rows [2].
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 d e f
## 3 h i j
tibble(x = c("a,b,c", "d,e", "f,g,i")) %>%
separate(x, c("um", "dois", "tres"))
## Warning: Expected 3 pieces. Missing pieces filled with `NA` in 1 rows [2].
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 d e <NA>
## 3 f g i
-
Tanto
unitecomoseparatepossuem um argumentoremove. Pra que ele serve e quando você o utilizaria no valorFALSE? -
Compare o argumento
values_fillempivot_widerefillemcomplete. Qual é a diferença?
Agradecimentos
Esse material é uma adaptação livre das vinhetas dos pacotes tidyr, readr e tibble e do capítulo Tidy Data do R for Data Science, de Wickham & Grolemund.