Soluções
Soluções dos exercícios.
readr, tibble, tidyr
# Não esqueça dos pacotes!
library(tidyverse)
## -- Attaching packages --------------------------------------- tidyverse 1.3.1 --
## v ggplot2 3.3.3 v purrr 0.3.4
## v tibble 3.1.2 v dplyr 1.0.6
## v tidyr 1.1.3 v stringr 1.4.0
## v readr 1.4.0 v forcats 0.5.1
## -- Conflicts ------------------------------------------ tidyverse_conflicts() --
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()
- Como você importaria o banco “epa78.csv”
file <- readr_example("epa78.txt")
Primeiro, é bom verificar como estão dispostas as informações no arquivo texto
read_lines(file, n_max = 10)
## [1] "ALFA ROMEO ALFA ROMEO 78010003"
## [2] "ALFETTA 03 81 8 74 7 89 9 ALFETTA 78010053"
## [3] "SPIDER 2000 01 SPIDER 2000 78010103"
## [4] "AMC AMC 78020002"
## [5] "GREMLIN 03 79 9 79 9 GREMLIN 78020053"
## [6] "PACER 04 89 11 89 11 PACER 78020103"
## [7] "PACER WAGON 07 90 26 91 26 PACER WAGON 78020153"
## [8] "CONCORD 04 88 12 90 11 90 11 83 16 CONCORD 78020203"
## [9] "CONCORD WAGON 07 91 30 91 30 CONCORD WAGON 78020253"
## [10] "MATADOR COUPE 05 97 14 97 14 MATADOR COUPE 78020303"
Ao identificar que se trata de um arquivo colunado, mas que as colunas são separadas por espaços, escolho o read_fwf com o fwf_empty.
dic <- fwf_empty(file)
df <- read_fwf(file, col_positions = dic)
##
## -- Column specification --------------------------------------------------------
## cols(
## X1 = col_character(),
## X2 = col_character(),
## X3 = col_double(),
## X4 = col_double(),
## X5 = col_double(),
## X6 = col_double(),
## X7 = col_double(),
## X8 = col_double(),
## X9 = col_double(),
## X10 = col_double(),
## X11 = col_character(),
## X12 = col_double()
## )
df
## # A tibble: 20 x 12
## X1 X2 X3 X4 X5 X6 X7 X8 X9 X10 X11 X12
## <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 ALFA RO~ <NA> NA NA NA NA NA NA NA NA ALFA R~ 7.80e7
## 2 ALFETTA 03 81 8 74 7 89 9 NA NA ALFETTA 7.80e7
## 3 SPIDER ~ 01 NA NA NA NA NA NA NA NA SPIDER~ 7.80e7
## 4 AMC <NA> NA NA NA NA NA NA NA NA AMC 7.80e7
## 5 GREMLIN 03 79 9 NA NA NA NA 79 9 GREMLIN 7.80e7
## 6 PACER 04 89 11 NA NA NA NA 89 11 PACER 7.80e7
## 7 PACER W~ 07 90 26 91 26 NA NA NA NA PACER ~ 7.80e7
## 8 CONCORD 04 88 12 90 11 90 11 83 16 CONCORD 7.80e7
## 9 CONCORD~ 07 91 30 NA NA 91 30 NA NA CONCOR~ 7.80e7
## 10 MATADOR~ 05 97 14 97 14 NA NA NA NA MATADO~ 7.80e7
## 11 MATADOR~ 06 110 20 NA NA 110 20 NA NA MATADO~ 7.80e7
## 12 MATADOR~ 09 112 50 NA NA 112 50 NA NA MATADO~ 7.80e7
## 13 ASTON M~ <NA> NA NA NA NA NA NA NA NA ASTON ~ 7.80e7
## 14 ASTON M~ <NA> NA NA NA NA NA NA NA NA ASTON ~ 7.80e7
## 15 AUDI <NA> NA NA NA NA NA NA NA NA AUDI 7.81e7
## 16 FOX 03 84 11 84 11 84 11 NA NA FOX 7.81e7
## 17 FOX WAG~ 07 83 40 NA NA 83 40 NA NA FOX WA~ 7.81e7
## 18 5000 04 90 15 NA NA 90 15 NA NA 5000 7.81e7
## 19 AVANTI <NA> NA NA NA NA NA NA NA NA AVANTI 7.81e7
## 20 AVANTI ~ 02 75 8 75 8 NA NA NA NA AVANTI~ 7.81e7
- Importe o banco “challenge.csv” e resolva os problemas com o tipo da coluna.
Ao verificar as primeiras 10 linhas do banco, podemos notar algo estranho
file <- readr_example("challenge.csv")
read_lines(file, n_max = 10)
## [1] "x,y" "404,NA" "4172,NA" "3004,NA" "787,NA" "37,NA" "2332,NA"
## [8] "2489,NA" "1449,NA" "3665,NA"
Parece ser um arquivo csv comum, com duas colunas, mas uma delas parece ter apenas NAs. Se a gente proceder com a importação padrão, chegaremos em
df <- read_csv(file)
##
## -- Column specification --------------------------------------------------------
## cols(
## x = col_double(),
## y = col_logical()
## )
## Warning: 1000 parsing failures.
## row col expected actual file
## 1001 y 1/0/T/F/TRUE/FALSE 2015-01-16 'C:/Users/vinic/Documents/R/win-library/4.1/readr/extdata/challenge.csv'
## 1002 y 1/0/T/F/TRUE/FALSE 2018-05-18 'C:/Users/vinic/Documents/R/win-library/4.1/readr/extdata/challenge.csv'
## 1003 y 1/0/T/F/TRUE/FALSE 2015-09-05 'C:/Users/vinic/Documents/R/win-library/4.1/readr/extdata/challenge.csv'
## 1004 y 1/0/T/F/TRUE/FALSE 2012-11-28 'C:/Users/vinic/Documents/R/win-library/4.1/readr/extdata/challenge.csv'
## 1005 y 1/0/T/F/TRUE/FALSE 2020-01-13 'C:/Users/vinic/Documents/R/win-library/4.1/readr/extdata/challenge.csv'
## .... ... .................. .......... ........................................................................
## See problems(...) for more details.
df
## # A tibble: 2,000 x 2
## x y
## <dbl> <lgl>
## 1 404 NA
## 2 4172 NA
## 3 3004 NA
## 4 787 NA
## 5 37 NA
## 6 2332 NA
## 7 2489 NA
## 8 1449 NA
## 9 3665 NA
## 10 3863 NA
## # ... with 1,990 more rows
No console de vocês, deve aparecer que foram importadas as colunas x como double e y como logical. Mas uma chuva de “parsing failures”, indicando que expected = 1/0/T/F/TRUE/FALSE, actual = 2015-01-16.
Na verdade, ao tentar adivinhar o tipo de colunas, o readr lê as primeiras 1000 observações em busca de um padrão. Você pode resolver esse problema:
# 1. Aumentando o número de observações utilizadas para adivinhar as colunas
df <- read_csv(file, guess_max = 1001)
##
## -- Column specification --------------------------------------------------------
## cols(
## x = col_double(),
## y = col_date(format = "")
## )
# A específicação da coluna Y agora é <date>
df
## # A tibble: 2,000 x 2
## x y
## <dbl> <date>
## 1 404 NA
## 2 4172 NA
## 3 3004 NA
## 4 787 NA
## 5 37 NA
## 6 2332 NA
## 7 2489 NA
## 8 1449 NA
## 9 3665 NA
## 10 3863 NA
## # ... with 1,990 more rows
# 2. escolhendo diretamente o tipo de coluna antes da importação
tipos <- cols(
y = col_date()
)
df <- read_csv(file, col_types = tipos)
# Mesmo resultado
df
## # A tibble: 2,000 x 2
## x y
## <dbl> <date>
## 1 404 NA
## 2 4172 NA
## 3 3004 NA
## 4 787 NA
## 5 37 NA
## 6 2332 NA
## 7 2489 NA
## 8 1449 NA
## 9 3665 NA
## 10 3863 NA
## # ... with 1,990 more rows
- Com o banco sala_aula dado a seguir, transforme-o para que ele contenha as variáveis nome, avaliação e nota.
sala_aula <- tribble(
~name, ~teste1, ~teste2, ~prova1,
"Billy", "<NA>", "D" , "C",
"Suzy", "F", "<NA>", "<NA>",
"Lionel", "B", "C" , "B",
"Jenny", "A", "A" , "B"
)
É sempre bom começar planejando o banco que queremos construir. Queremos um banco que tenha 3 variáveis: o nome, o tipo de prova aplicada e a nota de cada aluno. Para isso, precisamos colocar os nomes das colunas teste1, teste2 e prova1 numa variável e os valores das células em outra. Vamos chamar essas colunas de “avaliação” e “nota”, elas formam um par.
Agora vamos chamar pivot_wider e especificar esses argumentos.
sala_aula %>% pivot_longer(
# Primeiro, identificamos as colunas que serão modificadas
cols = c(teste1, teste2, prova1),
# Agora, indicamos os nomes das colunas que receberão
# os nomes das colunas transformadas
names_to = "avaliacao",
# os valores das células
values_to = "nota"
)
## # A tibble: 12 x 3
## name avaliacao nota
## <chr> <chr> <chr>
## 1 Billy teste1 <NA>
## 2 Billy teste2 D
## 3 Billy prova1 C
## 4 Suzy teste1 F
## 5 Suzy teste2 <NA>
## 6 Suzy prova1 <NA>
## 7 Lionel teste1 B
## 8 Lionel teste2 C
## 9 Lionel prova1 B
## 10 Jenny teste1 A
## 11 Jenny teste2 A
## 12 Jenny prova1 B
- Transforme o banco
relig_incomepara que ele contenha as colunas religião, renda e frequência.
relig_income
## # A tibble: 18 x 11
## religion `<$10k` `$10-20k` `$20-30k` `$30-40k` `$40-50k` `$50-75k` `$75-100k`
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Agnostic 27 34 60 81 76 137 122
## 2 Atheist 12 27 37 52 35 70 73
## 3 Buddhist 27 21 30 34 33 58 62
## 4 Catholic 418 617 732 670 638 1116 949
## 5 Don’t k~ 15 14 15 11 10 35 21
## 6 Evangel~ 575 869 1064 982 881 1486 949
## 7 Hindu 1 9 7 9 11 34 47
## 8 Histori~ 228 244 236 238 197 223 131
## 9 Jehovah~ 20 27 24 24 21 30 15
## 10 Jewish 19 19 25 25 30 95 69
## 11 Mainlin~ 289 495 619 655 651 1107 939
## 12 Mormon 29 40 48 51 56 112 85
## 13 Muslim 6 7 9 10 9 23 16
## 14 Orthodox 13 17 23 32 32 47 38
## 15 Other C~ 9 7 11 13 13 14 18
## 16 Other F~ 20 33 40 46 49 63 46
## 17 Other W~ 5 2 3 4 2 7 3
## 18 Unaffil~ 217 299 374 365 341 528 407
## # ... with 3 more variables: $100-150k <dbl>, >150k <dbl>,
## # Don't know/refused <dbl>
O banco relig_income parece ter uma organização em que temos 2 variáveis, mas uma delas está numa coluna “religion” e a outra está em 10 colunas, “income”. Queremos um banco que tenha 3 colunas: a religião, o nível de renda, e o número de pessoas em cada combinação das duas primeiras.
Como no exerício anterior, vamos chamar pivot_longer e especificar
relig_income %>% pivot_longer(
# As colunas a serem modificadas, notem o uso de ':' para selecionar várias
# colunas em sequência
cols = `<$10k`:`Don't know/refused`,
# Variável recebe os nomes da antiga coluna
names_to = "nivel_renda",
# Variável recebe os valores das células
values_to = "contagem"
)
## # A tibble: 180 x 3
## religion nivel_renda contagem
## <chr> <chr> <dbl>
## 1 Agnostic <$10k 27
## 2 Agnostic $10-20k 34
## 3 Agnostic $20-30k 60
## 4 Agnostic $30-40k 81
## 5 Agnostic $40-50k 76
## 6 Agnostic $50-75k 137
## 7 Agnostic $75-100k 122
## 8 Agnostic $100-150k 109
## 9 Agnostic >150k 84
## 10 Agnostic Don't know/refused 96
## # ... with 170 more rows
- Transforme o banco
billboardpara que ele contenha apenas uma coluna “semana” e uma coluna com a posição da música no ranking.
Dica, você pode selecionar várias colunas usando o atalho wk1:wk76
billboard
## # A tibble: 317 x 79
## artist track date.entered wk1 wk2 wk3 wk4 wk5 wk6 wk7 wk8
## <chr> <chr> <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2 Pac Baby D~ 2000-02-26 87 82 72 77 87 94 99 NA
## 2 2Ge+her The Ha~ 2000-09-02 91 87 92 NA NA NA NA NA
## 3 3 Doors~ Krypto~ 2000-04-08 81 70 68 67 66 57 54 53
## 4 3 Doors~ Loser 2000-10-21 76 76 72 69 67 65 55 59
## 5 504 Boyz Wobble~ 2000-04-15 57 34 25 17 17 31 36 49
## 6 98^0 Give M~ 2000-08-19 51 39 34 26 26 19 2 2
## 7 A*Teens Dancin~ 2000-07-08 97 97 96 95 100 NA NA NA
## 8 Aaliyah I Don'~ 2000-01-29 84 62 51 41 38 35 35 38
## 9 Aaliyah Try Ag~ 2000-03-18 59 53 38 28 21 18 16 14
## 10 Adams, ~ Open M~ 2000-08-26 76 76 74 69 68 67 61 58
## # ... with 307 more rows, and 68 more variables: wk9 <dbl>, wk10 <dbl>,
## # wk11 <dbl>, wk12 <dbl>, wk13 <dbl>, wk14 <dbl>, wk15 <dbl>, wk16 <dbl>,
## # wk17 <dbl>, wk18 <dbl>, wk19 <dbl>, wk20 <dbl>, wk21 <dbl>, wk22 <dbl>,
## # wk23 <dbl>, wk24 <dbl>, wk25 <dbl>, wk26 <dbl>, wk27 <dbl>, wk28 <dbl>,
## # wk29 <dbl>, wk30 <dbl>, wk31 <dbl>, wk32 <dbl>, wk33 <dbl>, wk34 <dbl>,
## # wk35 <dbl>, wk36 <dbl>, wk37 <dbl>, wk38 <dbl>, wk39 <dbl>, wk40 <dbl>,
## # wk41 <dbl>, wk42 <dbl>, wk43 <dbl>, wk44 <dbl>, wk45 <dbl>, wk46 <dbl>,
## # wk47 <dbl>, wk48 <dbl>, wk49 <dbl>, wk50 <dbl>, wk51 <dbl>, wk52 <dbl>,
## # wk53 <dbl>, wk54 <dbl>, wk55 <dbl>, wk56 <dbl>, wk57 <dbl>, wk58 <dbl>,
## # wk59 <dbl>, wk60 <dbl>, wk61 <dbl>, wk62 <dbl>, wk63 <dbl>, wk64 <dbl>,
## # wk65 <dbl>, wk66 <lgl>, wk67 <lgl>, wk68 <lgl>, wk69 <lgl>, wk70 <lgl>,
## # wk71 <lgl>, wk72 <lgl>, wk73 <lgl>, wk74 <lgl>, wk75 <lgl>, wk76 <lgl>
Da mesma forma como fizemos nos anteriores, queremos transformar as várias wk1:wk76 em um par de colunas, uma que me diga a semana e a outra que me diga em que posição no ranking a música estava.
billboard %>% pivot_longer(
# Colunas que serão transformadas
cols = wk1:wk76,
# Nome da variável que receberá os nomes das colunas
names_to = "semana",
# Nome da variável que receberá os valores das células
values_to = "posicao_rank",
# Nesse caso, uso o argumento opcional para eliminar os NAs
values_drop_na = TRUE
# Experimente mudar este argumento para FALSE e veja o resultado
# Quando uma música não está mais nas paradas, ela recebe NA. Acho
# justificado excluir os NAs nesse caso.
)
## # A tibble: 5,307 x 5
## artist track date.entered semana posicao_rank
## <chr> <chr> <date> <chr> <dbl>
## 1 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk1 87
## 2 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk2 82
## 3 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk3 72
## 4 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk4 77
## 5 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk5 87
## 6 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk6 94
## 7 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk7 99
## 8 2Ge+her The Hardest Part Of ... 2000-09-02 wk1 91
## 9 2Ge+her The Hardest Part Of ... 2000-09-02 wk2 87
## 10 2Ge+her The Hardest Part Of ... 2000-09-02 wk3 92
## # ... with 5,297 more rows
- Experimente fazer o caminho inverso dos exercícios 3 a 5, devolvendo os datasets ao seu formato original. O que você observou?
Vou começar enxugando os códigos anteriores para criar os resultados que produzimos e salvá-los em objetos.
sala_aula_long <-
sala_aula %>% pivot_longer(
cols = c(teste1, teste2, prova1),
names_to = "avaliacao",
values_to = "nota"
)
relig_income_long <-
relig_income %>% pivot_longer(
cols = `<$10k`:`Don't know/refused`,
names_to = "nivel_renda",
values_to = "contagem"
)
billboard_long <-
billboard %>% pivot_longer(
cols = wk1:wk76,
names_to = "semana",
values_to = "posicao_rank",
values_drop_na = TRUE
)
sala_aula_long
## # A tibble: 12 x 3
## name avaliacao nota
## <chr> <chr> <chr>
## 1 Billy teste1 <NA>
## 2 Billy teste2 D
## 3 Billy prova1 C
## 4 Suzy teste1 F
## 5 Suzy teste2 <NA>
## 6 Suzy prova1 <NA>
## 7 Lionel teste1 B
## 8 Lionel teste2 C
## 9 Lionel prova1 B
## 10 Jenny teste1 A
## 11 Jenny teste2 A
## 12 Jenny prova1 B
relig_income_long
## # A tibble: 180 x 3
## religion nivel_renda contagem
## <chr> <chr> <dbl>
## 1 Agnostic <$10k 27
## 2 Agnostic $10-20k 34
## 3 Agnostic $20-30k 60
## 4 Agnostic $30-40k 81
## 5 Agnostic $40-50k 76
## 6 Agnostic $50-75k 137
## 7 Agnostic $75-100k 122
## 8 Agnostic $100-150k 109
## 9 Agnostic >150k 84
## 10 Agnostic Don't know/refused 96
## # ... with 170 more rows
billboard_long
## # A tibble: 5,307 x 5
## artist track date.entered semana posicao_rank
## <chr> <chr> <date> <chr> <dbl>
## 1 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk1 87
## 2 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk2 82
## 3 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk3 72
## 4 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk4 77
## 5 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk5 87
## 6 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk6 94
## 7 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk7 99
## 8 2Ge+her The Hardest Part Of ... 2000-09-02 wk1 91
## 9 2Ge+her The Hardest Part Of ... 2000-09-02 wk2 87
## 10 2Ge+her The Hardest Part Of ... 2000-09-02 wk3 92
## # ... with 5,297 more rows
O caminho inverso desses bancos de dados, é utilizar pivot_wider. Aqui, vamos escolher um par de colunas que contém:
- O nome das colunas que queremos criar
- O valor que queremos passar para as células dessas novas colunas
Vamos ver exemplos comentados como no anterior
sala_aula_long %>% pivot_wider(
# Aqui, identificamos colunas que NÃO SERÃO MODIFICADAS
# É o contrário de pivot_longer. Por padrão, são todas as que não são
# mencionadas na transformação, mas para deixar bem claro,
# vou deixar explícito.
id_cols = name,
# Variável com os nomes para as novas colunas
names_from = avaliacao,
# Variável com os valores para as células
values_from = nota
)
## # A tibble: 4 x 4
## name teste1 teste2 prova1
## <chr> <chr> <chr> <chr>
## 1 Billy <NA> D C
## 2 Suzy F <NA> <NA>
## 3 Lionel B C B
## 4 Jenny A A B
relig_income_long %>% pivot_wider(
# Colunas não modificadas
id_cols = religion,
# Variável com os nomes para as novas colunas
names_from = nivel_renda,
# Variável com os valores para as células
values_from = contagem
)
## # A tibble: 18 x 11
## religion `<$10k` `$10-20k` `$20-30k` `$30-40k` `$40-50k` `$50-75k` `$75-100k`
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Agnostic 27 34 60 81 76 137 122
## 2 Atheist 12 27 37 52 35 70 73
## 3 Buddhist 27 21 30 34 33 58 62
## 4 Catholic 418 617 732 670 638 1116 949
## 5 Don’t k~ 15 14 15 11 10 35 21
## 6 Evangel~ 575 869 1064 982 881 1486 949
## 7 Hindu 1 9 7 9 11 34 47
## 8 Histori~ 228 244 236 238 197 223 131
## 9 Jehovah~ 20 27 24 24 21 30 15
## 10 Jewish 19 19 25 25 30 95 69
## 11 Mainlin~ 289 495 619 655 651 1107 939
## 12 Mormon 29 40 48 51 56 112 85
## 13 Muslim 6 7 9 10 9 23 16
## 14 Orthodox 13 17 23 32 32 47 38
## 15 Other C~ 9 7 11 13 13 14 18
## 16 Other F~ 20 33 40 46 49 63 46
## 17 Other W~ 5 2 3 4 2 7 3
## 18 Unaffil~ 217 299 374 365 341 528 407
## # ... with 3 more variables: $100-150k <dbl>, >150k <dbl>,
## # Don't know/refused <dbl>
billboard_long %>% pivot_wider(
# Colunas não modificadas
id_cols = c(artist, track, date.entered),
# Variável com os nomes para as novas colunas
names_from = semana,
# Variável com os valores para as células
values_from = posicao_rank
)
## # A tibble: 317 x 68
## artist track date.entered wk1 wk2 wk3 wk4 wk5 wk6 wk7 wk8
## <chr> <chr> <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2 Pac Baby D~ 2000-02-26 87 82 72 77 87 94 99 NA
## 2 2Ge+her The Ha~ 2000-09-02 91 87 92 NA NA NA NA NA
## 3 3 Doors~ Krypto~ 2000-04-08 81 70 68 67 66 57 54 53
## 4 3 Doors~ Loser 2000-10-21 76 76 72 69 67 65 55 59
## 5 504 Boyz Wobble~ 2000-04-15 57 34 25 17 17 31 36 49
## 6 98^0 Give M~ 2000-08-19 51 39 34 26 26 19 2 2
## 7 A*Teens Dancin~ 2000-07-08 97 97 96 95 100 NA NA NA
## 8 Aaliyah I Don'~ 2000-01-29 84 62 51 41 38 35 35 38
## 9 Aaliyah Try Ag~ 2000-03-18 59 53 38 28 21 18 16 14
## 10 Adams, ~ Open M~ 2000-08-26 76 76 74 69 68 67 61 58
## # ... with 307 more rows, and 57 more variables: wk9 <dbl>, wk10 <dbl>,
## # wk11 <dbl>, wk12 <dbl>, wk13 <dbl>, wk14 <dbl>, wk15 <dbl>, wk16 <dbl>,
## # wk17 <dbl>, wk18 <dbl>, wk19 <dbl>, wk20 <dbl>, wk21 <dbl>, wk22 <dbl>,
## # wk23 <dbl>, wk24 <dbl>, wk25 <dbl>, wk26 <dbl>, wk27 <dbl>, wk28 <dbl>,
## # wk29 <dbl>, wk30 <dbl>, wk31 <dbl>, wk32 <dbl>, wk33 <dbl>, wk34 <dbl>,
## # wk35 <dbl>, wk36 <dbl>, wk37 <dbl>, wk38 <dbl>, wk39 <dbl>, wk40 <dbl>,
## # wk41 <dbl>, wk42 <dbl>, wk43 <dbl>, wk44 <dbl>, wk45 <dbl>, wk46 <dbl>,
## # wk47 <dbl>, wk48 <dbl>, wk49 <dbl>, wk50 <dbl>, wk51 <dbl>, wk52 <dbl>,
## # wk53 <dbl>, wk54 <dbl>, wk55 <dbl>, wk56 <dbl>, wk57 <dbl>, wk58 <dbl>,
## # wk59 <dbl>, wk60 <dbl>, wk61 <dbl>, wk62 <dbl>, wk63 <dbl>, wk64 <dbl>,
## # wk65 <dbl>
Tanto pivot_longer quanto pivot_wider tem mais argumentos para lidar com situações complexas, como quando você precisa aplicar transformações em variáveis antes de reformatar o banco ou precisa utilizar múltiplas colunas, mas eu deixo isso para vocês descobrirem por conta própria quando estiverem confortáveis com a sintaxe básica.
- O que os argumentos
extraefillem separate fazem? Utilize o exemplo a seguir como guia.
tibble(x = c("a,b,c", "d,e,f,g", "h,i,j")) %>%
separate(x, c("um", "dois", "tres"))
## Warning: Expected 3 pieces. Additional pieces discarded in 1 rows [2].
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 d e f
## 3 h i j
tibble(x = c("a,b,c", "d,e", "f,g,i")) %>%
separate(x, c("um", "dois", "tres"))
## Warning: Expected 3 pieces. Missing pieces filled with `NA` in 1 rows [2].
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 d e <NA>
## 3 f g i
Por padrão, separate espera que todas as colunas sendo separadas tenham o mesmo comprimento. Por exemplo, no primeiro caso, indicamos que vamos criar três novas colunas, chamadas de “um”, “dois” e “tres”. Mas os vetores tem tamanhos diferentes. Um deles tem 4 letras ao invés de 3. No segundo exemplo, um deles tem duas letras ao invés de três. Esse tipo de situação é bastante comum quando lidamos com erros de digitação. Então, o que fazer com o elemento que está sobrando ou faltando?
# Sobrando
tibble(x = c("a,b,c", "d,e,f,g", "h,i,j")) %>%
separate(x, c("um", "dois", "tres"), extra = "warn") # avise que ocorreu (padrão)
## Warning: Expected 3 pieces. Additional pieces discarded in 1 rows [2].
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 d e f
## 3 h i j
tibble(x = c("a,b,c", "d,e,f,g", "h,i,j")) %>%
separate(x, c("um", "dois", "tres"), extra = "drop") # descarte o que sobrou
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 d e f
## 3 h i j
tibble(x = c("a,b,c", "d,e,f,g", "h,i,j")) %>%
separate(x, c("um", "dois", "tres"), extra = "merge") # junte com o último
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 d e f,g
## 3 h i j
# Note especialmente no último caso o que ocorreu com as colunas.
# Faltando
tibble(x = c("a,b,c", "d,e", "f,g,i")) %>%
separate(x, c("um", "dois", "tres"), fill = "warn") # avise e preencha a direita
## Warning: Expected 3 pieces. Missing pieces filled with `NA` in 1 rows [2].
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 d e <NA>
## 3 f g i
tibble(x = c("a,b,c", "d,e", "f,g,i")) %>%
separate(x, c("um", "dois", "tres"), fill = "left") # preencha a esquerda
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 <NA> d e
## 3 f g i
tibble(x = c("a,b,c", "d,e", "f,g,i")) %>%
separate(x, c("um", "dois", "tres"), fill = "right") # preencha a direta
## # A tibble: 3 x 3
## um dois tres
## <chr> <chr> <chr>
## 1 a b c
## 2 d e <NA>
## 3 f g i
# Note na sua saída do R como ficou a tibble e onde foram colocados NAs
# em cada caso
- Tanto
unitecomoseparatepossuem um argumentoremove. Pra que ele serve e quando você o utilizaria no valorFALSE?
Acho que a melhor forma de compreender isso é utilizar um banco de dados. Vamos pegar aquele da população retirado da Wikipédia.
populacao <- tribble(
~Rank, ~Country, ~Population, ~'% of world', ~Day, ~Month, ~Year, ~Source,
1L, "China", 1411778724, "17.9%", "1", "Nov", "2020", "Seventh Census on 2020",
2L, "India", 1377123716, "17.5%", "19", "May", "2021", "National population clock[3]",
3L, "United States", 331695937, "4.22%", "19", "May", "2021", "National population clock[4]",
4L, "Indonesia", 271350000, "3.45%", "31", "Dec", "2020", "National annual estimate[5]",
5L, "Pakistan", 225200000, "2.86%", "1", "Jul", "2021", "UN projection[2]",
6L, "Brazil", 213154869, "2.71%", "19", "May", "2021", "National population clock[6]",
7L, "Nigeria", 211401000, "2.69%", "1", "Jul", "2021", "UN projection[2]",
8L, "Bangladesh", 170689832, "2.17%", "19", "May", "2021", "National population clock[7]",
9L, "Russia", 146171015, "1.86%", "1", "Jan", "2021", "National annual estimate[8]",
10L, "Mexico", 126014024, "1.60%", "2", "Mar", "2020", "2020 census result[9]"
)
populacao
## # A tibble: 10 x 8
## Rank Country Population `% of world` Day Month Year Source
## <int> <chr> <dbl> <chr> <chr> <chr> <chr> <chr>
## 1 1 China 1411778724 17.9% 1 Nov 2020 Seventh Census o~
## 2 2 India 1377123716 17.5% 19 May 2021 National populat~
## 3 3 United Sta~ 331695937 4.22% 19 May 2021 National populat~
## 4 4 Indonesia 271350000 3.45% 31 Dec 2020 National annual ~
## 5 5 Pakistan 225200000 2.86% 1 Jul 2021 UN projection[2]
## 6 6 Brazil 213154869 2.71% 19 May 2021 National populat~
## 7 7 Nigeria 211401000 2.69% 1 Jul 2021 UN projection[2]
## 8 8 Bangladesh 170689832 2.17% 19 May 2021 National populat~
## 9 9 Russia 146171015 1.86% 1 Jan 2021 National annual ~
## 10 10 Mexico 126014024 1.60% 2 Mar 2020 2020 census resu~
Vamos ver dois exemplos, um com unite e outro com separate para exemplificar o que remove faz.
# Unir as colunas Day, Month, Year, remove = TRUE
populacao %>% unite(Data, Day, Month, Year, remove = TRUE) # padrão
## # A tibble: 10 x 6
## Rank Country Population `% of world` Data Source
## <int> <chr> <dbl> <chr> <chr> <chr>
## 1 1 China 1411778724 17.9% 1_Nov_2020 Seventh Census on 2020
## 2 2 India 1377123716 17.5% 19_May_20~ National population cl~
## 3 3 United Stat~ 331695937 4.22% 19_May_20~ National population cl~
## 4 4 Indonesia 271350000 3.45% 31_Dec_20~ National annual estima~
## 5 5 Pakistan 225200000 2.86% 1_Jul_2021 UN projection[2]
## 6 6 Brazil 213154869 2.71% 19_May_20~ National population cl~
## 7 7 Nigeria 211401000 2.69% 1_Jul_2021 UN projection[2]
## 8 8 Bangladesh 170689832 2.17% 19_May_20~ National population cl~
## 9 9 Russia 146171015 1.86% 1_Jan_2021 National annual estima~
## 10 10 Mexico 126014024 1.60% 2_Mar_2020 2020 census result[9]
# Unir as colunas Day, Month, Year, remove = FALSE
populacao %>% unite(Data, Day, Month, Year, remove = FALSE)
## # A tibble: 10 x 9
## Rank Country Population `% of world` Data Day Month Year Source
## <int> <chr> <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 1 China 1411778724 17.9% 1_Nov_~ 1 Nov 2020 Seventh Ce~
## 2 2 India 1377123716 17.5% 19_May~ 19 May 2021 National p~
## 3 3 United S~ 331695937 4.22% 19_May~ 19 May 2021 National p~
## 4 4 Indonesia 271350000 3.45% 31_Dec~ 31 Dec 2020 National a~
## 5 5 Pakistan 225200000 2.86% 1_Jul_~ 1 Jul 2021 UN project~
## 6 6 Brazil 213154869 2.71% 19_May~ 19 May 2021 National p~
## 7 7 Nigeria 211401000 2.69% 1_Jul_~ 1 Jul 2021 UN project~
## 8 8 Banglade~ 170689832 2.17% 19_May~ 19 May 2021 National p~
## 9 9 Russia 146171015 1.86% 1_Jan_~ 1 Jan 2021 National a~
## 10 10 Mexico 126014024 1.60% 2_Mar_~ 2 Mar 2020 2020 censu~
# Vejam o que aconteceu com as colunas nos dois bancos.
Agora com separate: Separar a coluna year em século e ano, apenas como exemplo
# remove = TRUE, padrão
populacao %>% separate(Year, c("seculo", "ano"), sep = 2, remove = TRUE)
## # A tibble: 10 x 9
## Rank Country Population `% of world` Day Month seculo ano Source
## <int> <chr> <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 1 China 1411778724 17.9% 1 Nov 20 20 Seventh Cen~
## 2 2 India 1377123716 17.5% 19 May 20 21 National po~
## 3 3 United S~ 331695937 4.22% 19 May 20 21 National po~
## 4 4 Indonesia 271350000 3.45% 31 Dec 20 20 National an~
## 5 5 Pakistan 225200000 2.86% 1 Jul 20 21 UN projecti~
## 6 6 Brazil 213154869 2.71% 19 May 20 21 National po~
## 7 7 Nigeria 211401000 2.69% 1 Jul 20 21 UN projecti~
## 8 8 Banglade~ 170689832 2.17% 19 May 20 21 National po~
## 9 9 Russia 146171015 1.86% 1 Jan 20 21 National an~
## 10 10 Mexico 126014024 1.60% 2 Mar 20 20 2020 census~
# remove = FALSE
populacao %>% separate(Year, c("seculo", "ano"), sep = 2, remove = FALSE)
## # A tibble: 10 x 10
## Rank Country Population `% of world` Day Month Year seculo ano Source
## <int> <chr> <dbl> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 1 China 1411778724 17.9% 1 Nov 2020 20 20 Sevent~
## 2 2 India 1377123716 17.5% 19 May 2021 20 21 Nation~
## 3 3 United ~ 331695937 4.22% 19 May 2021 20 21 Nation~
## 4 4 Indones~ 271350000 3.45% 31 Dec 2020 20 20 Nation~
## 5 5 Pakistan 225200000 2.86% 1 Jul 2021 20 21 UN pro~
## 6 6 Brazil 213154869 2.71% 19 May 2021 20 21 Nation~
## 7 7 Nigeria 211401000 2.69% 1 Jul 2021 20 21 UN pro~
## 8 8 Banglad~ 170689832 2.17% 19 May 2021 20 21 Nation~
## 9 9 Russia 146171015 1.86% 1 Jan 2021 20 21 Nation~
## 10 10 Mexico 126014024 1.60% 2 Mar 2020 20 20 2020 c~
# Vejam o que aconteceu com as colunas nos dois bancos.
Eu gosto de utilizar esse argumento quando eu tenho dúvida sobre o resultado e quero fazer inspeção visual para detectar eventuais problemas na separação ou junção. Uma vez que estou satisfeito com o resultado, em geral eu uso o remove=TRUE. Vocês tem que decidir se precisam manter as colunas originais ou se a coluna transformada é o suficiente.
- Compare o argumento
values_fillempivot_widerefillemcomplete. Qual é a diferença?
A resposta curta é simples: em pivot_wider, podemos ter aqueles missings “implícitos” que não apareciam no nosso banco longo e, durante a transformação, eles viram NAs nas colunas. Ó argumento values_fill indica um valor para ser preenchido no lugar de NA.
Em complete, temos uma situação similar. O que fazer quando for encontrada uma combinação de valores no banco longo que é um “missing implícito”? Você pode especificar um valor padrão para preenchê-lo.
São funções similares, mas uma funciona sem reformatar o banco e a outra durante o processo de reformatação. Veja um exemplo abaixo com aquela tibble das ações.
acoes <- tibble(
ano = c(2015, 2015, 2015, 2015, 2016, 2016, 2016),
qdr = c( 1, 2, 3, 4, 2, 3, 4),
lucro = c(1.88, 0.59, 0.35, NA, 0.92, 0.17, 2.66)
)
acoes
## # A tibble: 7 x 3
## ano qdr lucro
## <dbl> <dbl> <dbl>
## 1 2015 1 1.88
## 2 2015 2 0.59
## 3 2015 3 0.35
## 4 2015 4 NA
## 5 2016 2 0.92
## 6 2016 3 0.17
## 7 2016 4 2.66
# Vamos supor que o NA implícito significa que a empresa teve
# lucro = 0 naquele quadrimestre.
# pivot_wider, values_fill
acoes %>% pivot_wider(
id_cols = ano,
names_from = qdr,
values_from = lucro,
values_fill = 0
)
## # A tibble: 2 x 5
## ano `1` `2` `3` `4`
## <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2015 1.88 0.59 0.35 NA
## 2 2016 0 0.92 0.17 2.66
# complete, fill
acoes %>% complete(ano, qdr, fill = list(lucro = 0))
## # A tibble: 8 x 3
## ano qdr lucro
## <dbl> <dbl> <dbl>
## 1 2015 1 1.88
## 2 2015 2 0.59
## 3 2015 3 0.35
## 4 2015 4 0
## 5 2016 1 0
## 6 2016 2 0.92
## 7 2016 3 0.17
## 8 2016 4 2.66
Note o resultado. E note também que values_fill em pivot_wider é um pouco mais criterioso na hora de fazer as transformações.
stringr, forcats e dplyr
library(nycflights13)
-
Encontre os vôos que:
-
Atrasaram mais de duas horas
flights %>% filter(dep_delay > 120)
## # A tibble: 9,723 x 19
## year month day dep_time sched_dep_time dep_delay arr_time sched_arr_time
## <int> <int> <int> <int> <int> <dbl> <int> <int>
## 1 2013 1 1 848 1835 853 1001 1950
## 2 2013 1 1 957 733 144 1056 853
## 3 2013 1 1 1114 900 134 1447 1222
## 4 2013 1 1 1540 1338 122 2020 1825
## 5 2013 1 1 1815 1325 290 2120 1542
## 6 2013 1 1 1842 1422 260 1958 1535
## 7 2013 1 1 1856 1645 131 2212 2005
## 8 2013 1 1 1934 1725 129 2126 1855
## 9 2013 1 1 1938 1703 155 2109 1823
## 10 2013 1 1 1942 1705 157 2124 1830
## # ... with 9,713 more rows, and 11 more variables: arr_delay <dbl>,
## # carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, dest <chr>,
## # air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>
- Com destino a Houston (
IAHouHOU)
flights %>% filter(dest %in% c("IAH", "HOU"))
## # A tibble: 9,313 x 19
## year month day dep_time sched_dep_time dep_delay arr_time sched_arr_time
## <int> <int> <int> <int> <int> <dbl> <int> <int>
## 1 2013 1 1 517 515 2 830 819
## 2 2013 1 1 533 529 4 850 830
## 3 2013 1 1 623 627 -4 933 932
## 4 2013 1 1 728 732 -4 1041 1038
## 5 2013 1 1 739 739 0 1104 1038
## 6 2013 1 1 908 908 0 1228 1219
## 7 2013 1 1 1028 1026 2 1350 1339
## 8 2013 1 1 1044 1045 -1 1352 1351
## 9 2013 1 1 1114 900 134 1447 1222
## 10 2013 1 1 1205 1200 5 1503 1505
## # ... with 9,303 more rows, and 11 more variables: arr_delay <dbl>,
## # carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, dest <chr>,
## # air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>
- Operados pela United, American ou Delta
unique(flights$carrier)
## [1] "UA" "AA" "B6" "DL" "EV" "MQ" "US" "WN" "VX" "FL" "AS" "9E" "F9" "HA" "YV"
## [16] "OO"
flights %>% filter(carrier %in% c("UA", "AA", "DL"))
## # A tibble: 139,504 x 19
## year month day dep_time sched_dep_time dep_delay arr_time sched_arr_time
## <int> <int> <int> <int> <int> <dbl> <int> <int>
## 1 2013 1 1 517 515 2 830 819
## 2 2013 1 1 533 529 4 850 830
## 3 2013 1 1 542 540 2 923 850
## 4 2013 1 1 554 600 -6 812 837
## 5 2013 1 1 554 558 -4 740 728
## 6 2013 1 1 558 600 -2 753 745
## 7 2013 1 1 558 600 -2 924 917
## 8 2013 1 1 558 600 -2 923 937
## 9 2013 1 1 559 600 -1 941 910
## 10 2013 1 1 559 600 -1 854 902
## # ... with 139,494 more rows, and 11 more variables: arr_delay <dbl>,
## # carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, dest <chr>,
## # air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>
- Decolaram entre julho e setembro
flights %>% filter(between(month, 7, 9))
## # A tibble: 86,326 x 19
## year month day dep_time sched_dep_time dep_delay arr_time sched_arr_time
## <int> <int> <int> <int> <int> <dbl> <int> <int>
## 1 2013 7 1 1 2029 212 236 2359
## 2 2013 7 1 2 2359 3 344 344
## 3 2013 7 1 29 2245 104 151 1
## 4 2013 7 1 43 2130 193 322 14
## 5 2013 7 1 44 2150 174 300 100
## 6 2013 7 1 46 2051 235 304 2358
## 7 2013 7 1 48 2001 287 308 2305
## 8 2013 7 1 58 2155 183 335 43
## 9 2013 7 1 100 2146 194 327 30
## 10 2013 7 1 100 2245 135 337 135
## # ... with 86,316 more rows, and 11 more variables: arr_delay <dbl>,
## # carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, dest <chr>,
## # air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>
- Chegaram com mais de duas horas de atraso, mas não decolaram com atraso
flights %>% filter(arr_delay > 120, dep_delay <= 0)
## # A tibble: 29 x 19
## year month day dep_time sched_dep_time dep_delay arr_time sched_arr_time
## <int> <int> <int> <int> <int> <dbl> <int> <int>
## 1 2013 1 27 1419 1420 -1 1754 1550
## 2 2013 10 7 1350 1350 0 1736 1526
## 3 2013 10 7 1357 1359 -2 1858 1654
## 4 2013 10 16 657 700 -3 1258 1056
## 5 2013 11 1 658 700 -2 1329 1015
## 6 2013 3 18 1844 1847 -3 39 2219
## 7 2013 4 17 1635 1640 -5 2049 1845
## 8 2013 4 18 558 600 -2 1149 850
## 9 2013 4 18 655 700 -5 1213 950
## 10 2013 5 22 1827 1830 -3 2217 2010
## # ... with 19 more rows, and 11 more variables: arr_delay <dbl>, carrier <chr>,
## # flight <int>, tailnum <chr>, origin <chr>, dest <chr>, air_time <dbl>,
## # distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>
- Atrasaram mais de uma hora para decolar, mas recuperaram mais de 30 minutos durante o voo
flights %>% filter(dep_delay > 60, dep_delay - arr_delay >= 30)
## # A tibble: 2,046 x 19
## year month day dep_time sched_dep_time dep_delay arr_time sched_arr_time
## <int> <int> <int> <int> <int> <dbl> <int> <int>
## 1 2013 1 1 1716 1545 91 2140 2039
## 2 2013 1 1 2205 1720 285 46 2040
## 3 2013 1 1 2326 2130 116 131 18
## 4 2013 1 3 1503 1221 162 1803 1555
## 5 2013 1 3 1821 1530 171 2131 1910
## 6 2013 1 3 1839 1700 99 2056 1950
## 7 2013 1 3 1850 1745 65 2148 2120
## 8 2013 1 3 1923 1815 68 2036 1958
## 9 2013 1 3 1941 1759 102 2246 2139
## 10 2013 1 3 1950 1845 65 2228 2227
## # ... with 2,036 more rows, and 11 more variables: arr_delay <dbl>,
## # carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, dest <chr>,
## # air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>
- Decolaram entre a meia-noite e 6 da manhã (inclusive)
flights %>% filter(between(hour, 0, 5) | (hour == 6 & minute == 0))
## # A tibble: 8,970 x 19
## year month day dep_time sched_dep_time dep_delay arr_time sched_arr_time
## <int> <int> <int> <int> <int> <dbl> <int> <int>
## 1 2013 1 1 517 515 2 830 819
## 2 2013 1 1 533 529 4 850 830
## 3 2013 1 1 542 540 2 923 850
## 4 2013 1 1 544 545 -1 1004 1022
## 5 2013 1 1 554 600 -6 812 837
## 6 2013 1 1 554 558 -4 740 728
## 7 2013 1 1 555 600 -5 913 854
## 8 2013 1 1 557 600 -3 709 723
## 9 2013 1 1 557 600 -3 838 846
## 10 2013 1 1 558 600 -2 753 745
## # ... with 8,960 more rows, and 11 more variables: arr_delay <dbl>,
## # carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, dest <chr>,
## # air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>
- Reordene suas colunas para encontrar os voos mais rápidos (maior velocidade de voo).
flights %>%
select(air_time, distance) %>%
mutate(speed = distance/air_time) %>%
arrange(desc(speed))
## # A tibble: 336,776 x 3
## air_time distance speed
## <dbl> <dbl> <dbl>
## 1 65 762 11.7
## 2 93 1008 10.8
## 3 55 594 10.8
## 4 70 748 10.7
## 5 105 1035 9.86
## 6 170 1598 9.4
## 7 172 1598 9.29
## 8 175 1623 9.27
## 9 173 1598 9.24
## 10 173 1598 9.24
## # ... with 336,766 more rows
- Teste várias maneiras diferentes de selecionar as variáveis
dep_time,dep_delay,arr_timeearr_delayusando as várias helper functions deselect.
flights %>% select(dep_time, dep_delay, arr_time, arr_delay)
## # A tibble: 336,776 x 4
## dep_time dep_delay arr_time arr_delay
## <int> <dbl> <int> <dbl>
## 1 517 2 830 11
## 2 533 4 850 20
## 3 542 2 923 33
## 4 544 -1 1004 -18
## 5 554 -6 812 -25
## 6 554 -4 740 12
## 7 555 -5 913 19
## 8 557 -3 709 -14
## 9 557 -3 838 -8
## 10 558 -2 753 8
## # ... with 336,766 more rows
flights %>% select(starts_with("dep"), starts_with("arr"))
## # A tibble: 336,776 x 4
## dep_time dep_delay arr_time arr_delay
## <int> <dbl> <int> <dbl>
## 1 517 2 830 11
## 2 533 4 850 20
## 3 542 2 923 33
## 4 544 -1 1004 -18
## 5 554 -6 812 -25
## 6 554 -4 740 12
## 7 555 -5 913 19
## 8 557 -3 709 -14
## 9 557 -3 838 -8
## 10 558 -2 753 8
## # ... with 336,766 more rows
flights %>% select(starts_with(c("dep", "arr")))
## # A tibble: 336,776 x 4
## dep_time dep_delay arr_time arr_delay
## <int> <dbl> <int> <dbl>
## 1 517 2 830 11
## 2 533 4 850 20
## 3 542 2 923 33
## 4 544 -1 1004 -18
## 5 554 -6 812 -25
## 6 554 -4 740 12
## 7 555 -5 913 19
## 8 557 -3 709 -14
## 9 557 -3 838 -8
## 10 558 -2 753 8
## # ... with 336,766 more rows
flights %>% select(matches("^arr|^dep"))
## # A tibble: 336,776 x 4
## dep_time dep_delay arr_time arr_delay
## <int> <dbl> <int> <dbl>
## 1 517 2 830 11
## 2 533 4 850 20
## 3 542 2 923 33
## 4 544 -1 1004 -18
## 5 554 -6 812 -25
## 6 554 -4 740 12
## 7 555 -5 913 19
## 8 557 -3 709 -14
## 9 557 -3 838 -8
## 10 558 -2 753 8
## # ... with 336,766 more rows
flights %>% select(!starts_with(c("sched", "car")) & contains(c("dep", "arr")))
## # A tibble: 336,776 x 4
## dep_time dep_delay arr_time arr_delay
## <int> <dbl> <int> <dbl>
## 1 517 2 830 11
## 2 533 4 850 20
## 3 542 2 923 33
## 4 544 -1 1004 -18
## 5 554 -6 812 -25
## 6 554 -4 740 12
## 7 555 -5 913 19
## 8 557 -3 709 -14
## 9 557 -3 838 -8
## 10 558 -2 753 8
## # ... with 336,766 more rows
flights %>% select(ends_with(c("time", "delay")) & !starts_with(c("sched", "air")))
## # A tibble: 336,776 x 4
## dep_time arr_time dep_delay arr_delay
## <int> <int> <dbl> <dbl>
## 1 517 830 2 11
## 2 533 850 4 20
## 3 542 923 2 33
## 4 544 1004 -1 -18
## 5 554 812 -6 -25
## 6 554 740 -4 12
## 7 555 913 -5 19
## 8 557 709 -3 -14
## 9 557 838 -3 -8
## 10 558 753 -2 8
## # ... with 336,766 more rows
- As variáveis
dep_timeesched_dep_timeestão num formato incorreto (veja?flights). Converta-as commutatepara um valor em minutos passados desde a meia-noite. Dica: utilize%/%e%%.
flights %>% mutate(
dep_hour = dep_time %/% 100,
dep_minute = dep_time %% 100,
sched_dep_hour = sched_dep_time %/% 100,
sched_arr_minute = sched_arr_time %% 100
)
## # A tibble: 336,776 x 23
## year month day dep_time sched_dep_time dep_delay arr_time sched_arr_time
## <int> <int> <int> <int> <int> <dbl> <int> <int>
## 1 2013 1 1 517 515 2 830 819
## 2 2013 1 1 533 529 4 850 830
## 3 2013 1 1 542 540 2 923 850
## 4 2013 1 1 544 545 -1 1004 1022
## 5 2013 1 1 554 600 -6 812 837
## 6 2013 1 1 554 558 -4 740 728
## 7 2013 1 1 555 600 -5 913 854
## 8 2013 1 1 557 600 -3 709 723
## 9 2013 1 1 557 600 -3 838 846
## 10 2013 1 1 558 600 -2 753 745
## # ... with 336,766 more rows, and 15 more variables: arr_delay <dbl>,
## # carrier <chr>, flight <int>, tailnum <chr>, origin <chr>, dest <chr>,
## # air_time <dbl>, distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>,
## # dep_hour <dbl>, dep_minute <dbl>, sched_dep_hour <dbl>,
## # sched_arr_minute <dbl>
# Há uma outra solução com separate!
flights %>%
separate(
col = dep_time,
into = c("dep_hour", "dep_minute"),
sep = 1,
# Esse argumento é importante! Teste com FALSE para ver a diferença
convert = TRUE) %>%
separate(
col = sched_dep_time,
into = c("sched_dep_hour", "sched_dep_minute"),
sep = 1,
convert = TRUE)
## # A tibble: 336,776 x 21
## year month day dep_hour dep_minute sched_dep_hour sched_dep_minute
## <int> <int> <int> <int> <int> <int> <int>
## 1 2013 1 1 5 17 5 15
## 2 2013 1 1 5 33 5 29
## 3 2013 1 1 5 42 5 40
## 4 2013 1 1 5 44 5 45
## 5 2013 1 1 5 54 6 0
## 6 2013 1 1 5 54 5 58
## 7 2013 1 1 5 55 6 0
## 8 2013 1 1 5 57 6 0
## 9 2013 1 1 5 57 6 0
## 10 2013 1 1 5 58 6 0
## # ... with 336,766 more rows, and 14 more variables: dep_delay <dbl>,
## # arr_time <int>, sched_arr_time <int>, arr_delay <dbl>, carrier <chr>,
## # flight <int>, tailnum <chr>, origin <chr>, dest <chr>, air_time <dbl>,
## # distance <dbl>, hour <dbl>, minute <dbl>, time_hour <dttm>
Existe uma outra solução possível para essa questão usando manipulação de strings, com str_sub também. Fica como desafio!
Pensando na legibilidade do código e na flexibilidade da abordagem, qual das duas soluções acima você implementaria? mutate ou duas separate? Reflita.
- O que o código abaixo está fazendo? Porque mesmo após o código abaixo continuam existindo diferenças entre os valores das variáveis
air_timeetravel_time?
flights %>%
select(air_time, dep_time, arr_time, dep_delay, arr_delay) %>%
mutate(dep_hour = dep_time %/% 100,
dep_min = dep_time %% 100,
dep_time2 = dep_hour * 60 + dep_min,
arr_hour = arr_time %/% 100,
arr_min = arr_time %% 100,
arr_time2 = arr_hour * 60 + arr_min,
travel_time = arr_time2 - dep_time2) %>%
select(-dep_hour, -dep_min, -arr_hour, -arr_min)
## # A tibble: 336,776 x 8
## air_time dep_time arr_time dep_delay arr_delay dep_time2 arr_time2
## <dbl> <int> <int> <dbl> <dbl> <dbl> <dbl>
## 1 227 517 830 2 11 317 510
## 2 227 533 850 4 20 333 530
## 3 160 542 923 2 33 342 563
## 4 183 544 1004 -1 -18 344 604
## 5 116 554 812 -6 -25 354 492
## 6 150 554 740 -4 12 354 460
## 7 158 555 913 -5 19 355 553
## 8 53 557 709 -3 -14 357 429
## 9 140 557 838 -3 -8 357 518
## 10 138 558 753 -2 8 358 473
## # ... with 336,766 more rows, and 1 more variable: travel_time <dbl>
Essa tem uma resposta mais qualitativa. A primeira parte é parecida com a questão anterior, mas estamos manualmente tentando calcular os tempos de viagem. Acontece que os valores não batem com os tempos de vôo identificados no banco. Isso se deve a pelo menos três questões distintas.
- Uma delas diz respeito ao registro dos tempos, a definição de
air_timepode não estar considerando tempos em que o avião está manobrando ou em solo ou mesmo podem existir erros de preenchimento. - A segunda diz respeito ao fuso horário distinto entre aeroportos de saída e chegada, que complica o cálculo dos tempos reais, então nosso cálculo está muito cru para identificar isso.
- A última questão são os vôos longos, que começam em um dia e terminam no dia seguinte, que podem prejudicar nosso método de cálculo. Para corrigir alguns desses problemas, você precisaria escrever um código que minimamente levasse essas questões em consideração. Como esse não é o objetivo do curso, eu deixo para quem quiser tentar. Há uma solução postada aqui.
- Use o stringr para concatenar as seguintes strings em uma frase
x <- "."
y <- "feliz"
w <- "acordei"
z <- "hoje"
str_c(z, w, y, sep = " ") %>%
str_c(x, sep = "") %>%
str_to_sentence()
## [1] "Hoje acordei feliz."
- Corrija as inconsistências nas colunas país, primeiro_nome, segundo_nome e crie uma nova coluna nomes contendo as duas anteriores. No final, ordene o banco em ordem alfabética.
df <-
tibble::tribble(
~pais, ~primeiro_nome, ~segundo_nome,
# -------|----------------|-------------|
"BRASIL", "ISABELA", "MARTINS",
"Brasil", "Eduardo", "cabellos",
"brasil", "márcia", "pinto",
"bRaSiL", "rogério", "Marinho",
)
# Sem dplyr
df$pais <- str_to_title(df$pais)
df$primeiro_nome <- str_to_title(df$primeiro_nome)
df$segundo_nome <- str_to_title(df$segundo_nome)
df <- df %>% tidyr::unite(nomes, primeiro_nome, segundo_nome, sep = " ")
df[ str_order(df$nomes), ]
## # A tibble: 4 x 2
## pais nomes
## <chr> <chr>
## 1 Brasil Eduardo Cabellos
## 2 Brasil Isabela Martins
## 3 Brasil Márcia Pinto
## 4 Brasil Rogério Marinho
# Com dplyr
df <-
tibble::tribble(
~pais, ~primeiro_nome, ~segundo_nome,
# -------|----------------|-------------|
"BRASIL", "ISABELA", "MARTINS",
"Brasil", "Eduardo", "cabellos",
"brasil", "márcia", "pinto",
"bRaSiL", "rogério", "Marinho",
)
df %>%
mutate(pais = str_to_title(pais),
primeiro_nome = str_to_title(primeiro_nome),
segundo_nome = str_to_title(segundo_nome)) %>%
unite(nomes, primeiro_nome, segundo_nome, sep = " ") %>%
arrange(str_order(nomes))
## # A tibble: 4 x 2
## pais nomes
## <chr> <chr>
## 1 Brasil Eduardo Cabellos
## 2 Brasil Isabela Martins
## 3 Brasil Márcia Pinto
## 4 Brasil Rogério Marinho
- Transforme a string
c("Seu nome", "Seu sobrenome da mãe", "Seu sobrenome do pai")na string"SEU SOBRENOME DO PAI, sua inicial do nome. sua inicial da mãe.", como numa citação. Veja o exemplo abaixo:
# Transforme
c("Vinícius", "de Souza", "Maia")
## [1] "Vinícius" "de Souza" "Maia"
# Resultado
"MAIA, V. S."
## [1] "MAIA, V. S."
x <- c("Vinícius", "de Souza", "Maia")
x[1] <- str_sub(x[1], 1, 1) %>% str_c(".")
x[2] <- str_sub(x[2], 4, 4) %>% str_c(".")
x[3] <- str_to_upper(x[3])
str_c(c(x[3], x[1], x[2]), collapse = " ")
## [1] "MAIA V. S."
- DESAFIO: Nos microdados da área de saúde, é comum que a variável idade esteja registrada da seguinte forma: “150”, “219”, “312”, “471”. Esses códigos indicam primeiro qual a unidade de medida da idade e segundo o valor desta unidade, 1 = horas, 2 = dias, 3 = meses, 4 = anos. Proponha um código usando
stringrpara transformar o vetor abaixo em um valor numérico.
# Não precisa se preocupar com essa parte
x <- as.character(round(c(
runif(25, 100, 124),
runif(25, 201, 230),
runif(25, 301, 312),
runif(25, 401, 499)
)))
# Como você transformaria esse vetor em número?
x
## [1] "118" "114" "118" "120" "109" "104" "114" "117" "122" "109" "110" "113"
## [13] "106" "100" "122" "121" "124" "124" "110" "100" "117" "119" "105" "118"
## [25] "122" "201" "205" "212" "216" "228" "213" "227" "221" "214" "227" "206"
## [37] "207" "215" "224" "223" "203" "221" "215" "225" "210" "208" "219" "207"
## [49] "221" "227" "310" "305" "301" "304" "310" "307" "305" "309" "308" "306"
## [61] "308" "305" "304" "303" "306" "302" "310" "306" "301" "310" "306" "304"
## [73] "305" "304" "307" "401" "459" "429" "435" "435" "415" "475" "414" "419"
## [85] "495" "475" "431" "460" "478" "447" "436" "412" "403" "423" "467" "438"
## [97] "475" "467" "418" "476"
# Esse exercício é um pouco mais difícil mesmo!
x %>% str_extract("\\d")
## [1] "1" "1" "1" "1" "1" "1" "1" "1" "1" "1" "1" "1" "1" "1" "1" "1" "1" "1"
## [19] "1" "1" "1" "1" "1" "1" "1" "2" "2" "2" "2" "2" "2" "2" "2" "2" "2" "2"
## [37] "2" "2" "2" "2" "2" "2" "2" "2" "2" "2" "2" "2" "2" "2" "3" "3" "3" "3"
## [55] "3" "3" "3" "3" "3" "3" "3" "3" "3" "3" "3" "3" "3" "3" "3" "3" "3" "3"
## [73] "3" "3" "3" "4" "4" "4" "4" "4" "4" "4" "4" "4" "4" "4" "4" "4" "4" "4"
## [91] "4" "4" "4" "4" "4" "4" "4" "4" "4" "4"
tibble(
tipo_idade = str_sub(x, 1, 1),
idade = str_sub(x, 2, 3),
idade_anos =
if_else(
str_detect(tipo_idade, "1"),
as.numeric(idade) / (24 * 30 * 12),
if_else(
str_detect(tipo_idade, "2"),
as.numeric(idade) / (30 * 12),
if_else(
str_detect(tipo_idade, "3"),
as.numeric(idade) / 12,
as.numeric(idade)
)
)
)
) %>%
print(n = Inf)
## # A tibble: 100 x 3
## tipo_idade idade idade_anos
## <chr> <chr> <dbl>
## 1 1 18 0.00208
## 2 1 14 0.00162
## 3 1 18 0.00208
## 4 1 20 0.00231
## 5 1 09 0.00104
## 6 1 04 0.000463
## 7 1 14 0.00162
## 8 1 17 0.00197
## 9 1 22 0.00255
## 10 1 09 0.00104
## 11 1 10 0.00116
## 12 1 13 0.00150
## 13 1 06 0.000694
## 14 1 00 0
## 15 1 22 0.00255
## 16 1 21 0.00243
## 17 1 24 0.00278
## 18 1 24 0.00278
## 19 1 10 0.00116
## 20 1 00 0
## 21 1 17 0.00197
## 22 1 19 0.00220
## 23 1 05 0.000579
## 24 1 18 0.00208
## 25 1 22 0.00255
## 26 2 01 0.00278
## 27 2 05 0.0139
## 28 2 12 0.0333
## 29 2 16 0.0444
## 30 2 28 0.0778
## 31 2 13 0.0361
## 32 2 27 0.075
## 33 2 21 0.0583
## 34 2 14 0.0389
## 35 2 27 0.075
## 36 2 06 0.0167
## 37 2 07 0.0194
## 38 2 15 0.0417
## 39 2 24 0.0667
## 40 2 23 0.0639
## 41 2 03 0.00833
## 42 2 21 0.0583
## 43 2 15 0.0417
## 44 2 25 0.0694
## 45 2 10 0.0278
## 46 2 08 0.0222
## 47 2 19 0.0528
## 48 2 07 0.0194
## 49 2 21 0.0583
## 50 2 27 0.075
## 51 3 10 0.833
## 52 3 05 0.417
## 53 3 01 0.0833
## 54 3 04 0.333
## 55 3 10 0.833
## 56 3 07 0.583
## 57 3 05 0.417
## 58 3 09 0.75
## 59 3 08 0.667
## 60 3 06 0.5
## 61 3 08 0.667
## 62 3 05 0.417
## 63 3 04 0.333
## 64 3 03 0.25
## 65 3 06 0.5
## 66 3 02 0.167
## 67 3 10 0.833
## 68 3 06 0.5
## 69 3 01 0.0833
## 70 3 10 0.833
## 71 3 06 0.5
## 72 3 04 0.333
## 73 3 05 0.417
## 74 3 04 0.333
## 75 3 07 0.583
## 76 4 01 1
## 77 4 59 59
## 78 4 29 29
## 79 4 35 35
## 80 4 35 35
## 81 4 15 15
## 82 4 75 75
## 83 4 14 14
## 84 4 19 19
## 85 4 95 95
## 86 4 75 75
## 87 4 31 31
## 88 4 60 60
## 89 4 78 78
## 90 4 47 47
## 91 4 36 36
## 92 4 12 12
## 93 4 03 3
## 94 4 23 23
## 95 4 67 67
## 96 4 38 38
## 97 4 75 75
## 98 4 67 67
## 99 4 18 18
## 100 4 76 76
Ao invés de utilizar essas chamadas recursivas de if_else, que são muito ruins de ler, como você poderia reescrever a condição usando case_when?
- Explore as contagens da variável
rincomeemgss_cat, ela ficaria bem representada num gráfico? De qual tipo?
gss_cat %>% count(rincome)
## # A tibble: 16 x 2
## rincome n
## <fct> <int>
## 1 No answer 183
## 2 Don't know 267
## 3 Refused 975
## 4 $25000 or more 7363
## 5 $20000 - 24999 1283
## 6 $15000 - 19999 1048
## 7 $10000 - 14999 1168
## 8 $8000 to 9999 340
## 9 $7000 to 7999 188
## 10 $6000 to 6999 215
## 11 $5000 to 5999 227
## 12 $4000 to 4999 226
## 13 $3000 to 3999 276
## 14 $1000 to 2999 395
## 15 Lt $1000 286
## 16 Not applicable 7043
Em geral, contagens de variáveis ficam bem em gráficos de barras ou visualizações equivalentes, em que é possível comparar visualmente as contagens das diversas categorias. Mais sobre isso na aula do ggplot2.
- Qual a religião mais comum em
gss_cat? Qual o partido (partyid) mais popular?
# Religião
gss_cat %>% count(relig) %>% arrange(desc(n))
## # A tibble: 15 x 2
## relig n
## <fct> <int>
## 1 Protestant 10846
## 2 Catholic 5124
## 3 None 3523
## 4 Christian 689
## 5 Jewish 388
## 6 Other 224
## 7 Buddhism 147
## 8 Inter-nondenominational 109
## 9 Moslem/islam 104
## 10 Orthodox-christian 95
## 11 No answer 93
## 12 Hinduism 71
## 13 Other eastern 32
## 14 Native american 23
## 15 Don't know 15
# Partido
gss_cat %>% count(partyid) %>% arrange(desc(n))
## # A tibble: 10 x 2
## partyid n
## <fct> <int>
## 1 Independent 4119
## 2 Not str democrat 3690
## 3 Strong democrat 3490
## 4 Not str republican 3032
## 5 Ind,near dem 2499
## 6 Strong republican 2314
## 7 Ind,near rep 1791
## 8 Other party 393
## 9 No answer 154
## 10 Don't know 1
- A que religião se refere a variável
denom? Você pode descobrir isso fazendo uma tabela de contagens?
Você pode chamar count com várias variáveis para fazer uma tabulação cruzada.
gss_cat %>% count(relig, denom) %>% print(n = Inf)
## # A tibble: 47 x 3
## relig denom n
## <fct> <fct> <int>
## 1 No answer No answer 93
## 2 Don't know Not applicable 15
## 3 Inter-nondenominational Not applicable 109
## 4 Native american Not applicable 23
## 5 Christian No answer 2
## 6 Christian Don't know 11
## 7 Christian No denomination 452
## 8 Christian Not applicable 224
## 9 Orthodox-christian Not applicable 95
## 10 Moslem/islam Not applicable 104
## 11 Other eastern Not applicable 32
## 12 Hinduism Not applicable 71
## 13 Buddhism Not applicable 147
## 14 Other No denomination 7
## 15 Other Not applicable 217
## 16 None Not applicable 3523
## 17 Jewish Not applicable 388
## 18 Catholic Not applicable 5124
## 19 Protestant No answer 22
## 20 Protestant Don't know 41
## 21 Protestant No denomination 1224
## 22 Protestant Other 2534
## 23 Protestant Episcopal 397
## 24 Protestant Presbyterian-dk wh 244
## 25 Protestant Presbyterian, merged 67
## 26 Protestant Other presbyterian 47
## 27 Protestant United pres ch in us 110
## 28 Protestant Presbyterian c in us 104
## 29 Protestant Lutheran-dk which 267
## 30 Protestant Evangelical luth 122
## 31 Protestant Other lutheran 30
## 32 Protestant Wi evan luth synod 71
## 33 Protestant Lutheran-mo synod 212
## 34 Protestant Luth ch in america 71
## 35 Protestant Am lutheran 146
## 36 Protestant Methodist-dk which 239
## 37 Protestant Other methodist 33
## 38 Protestant United methodist 1067
## 39 Protestant Afr meth ep zion 32
## 40 Protestant Afr meth episcopal 77
## 41 Protestant Baptist-dk which 1457
## 42 Protestant Other baptists 213
## 43 Protestant Southern baptist 1536
## 44 Protestant Nat bapt conv usa 40
## 45 Protestant Nat bapt conv of am 76
## 46 Protestant Am bapt ch in usa 130
## 47 Protestant Am baptist asso 237
- Como você poderia diminuir o número de categorias da variável
rincomedo bancogss_cat?
A melhor função para redução de fatores é fct_collapse. Veja como ficam a coluna original e a transformada.
gss_cat2 <-
gss_cat %>%
# Aqui vou salvar em "rincome2" para a gente poder ver as duas
mutate(rincome2 = fct_collapse(
rincome,
"Non-response" = c("No answer", "Don't know", "Refused", "Not applicable"),
"Até 5k" = c("$4000 to 4999", "$3000 to 3999", "$1000 to 2999", "Lt $1000"),
"5k-10k" = c( "$8000 to 9999", "$7000 to 7999", "$6000 to 6999", "$5000 to 5999"),
"10k-20k" = c("$15000 - 19999", "$10000 - 14999"),
"20k+" = c("$25000 or more", "$20000 - 24999"))) %>%
select(rincome, rincome2)
# E veja as contagens
gss_cat2 %>% count(rincome)
## # A tibble: 16 x 2
## rincome n
## <fct> <int>
## 1 No answer 183
## 2 Don't know 267
## 3 Refused 975
## 4 $25000 or more 7363
## 5 $20000 - 24999 1283
## 6 $15000 - 19999 1048
## 7 $10000 - 14999 1168
## 8 $8000 to 9999 340
## 9 $7000 to 7999 188
## 10 $6000 to 6999 215
## 11 $5000 to 5999 227
## 12 $4000 to 4999 226
## 13 $3000 to 3999 276
## 14 $1000 to 2999 395
## 15 Lt $1000 286
## 16 Not applicable 7043
gss_cat2 %>% count(rincome2)
## # A tibble: 5 x 2
## rincome2 n
## <fct> <int>
## 1 Non-response 8468
## 2 20k+ 8646
## 3 10k-20k 2216
## 4 5k-10k 970
## 5 Até 5k 1183
ggplot2
- O que tem de errado no código abaixo? Por que os pontos não ficaram azuis?
ggplot(data = mpg) +
geom_point(mapping = aes(x = displ, y = hwy, color = "blue"))
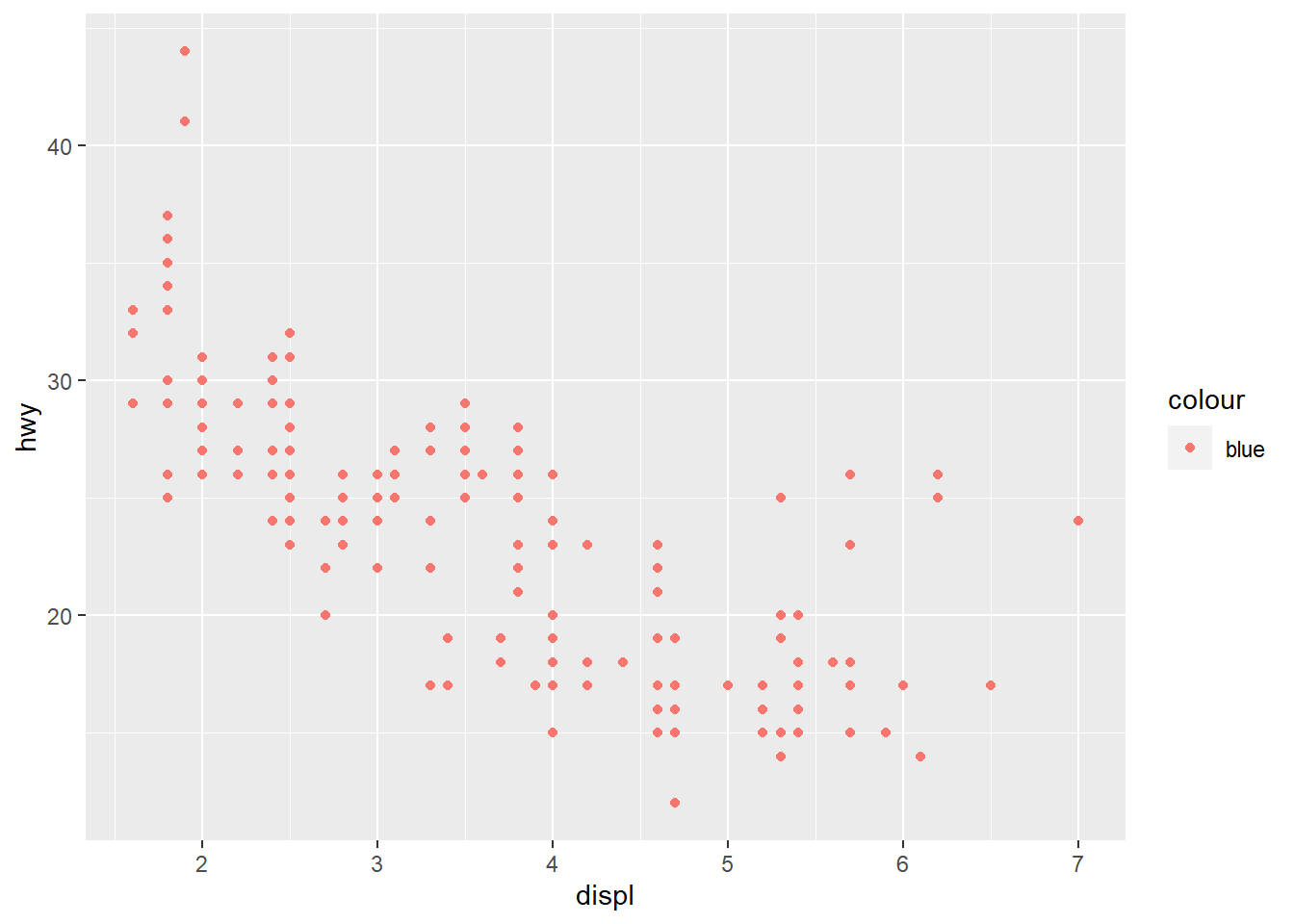
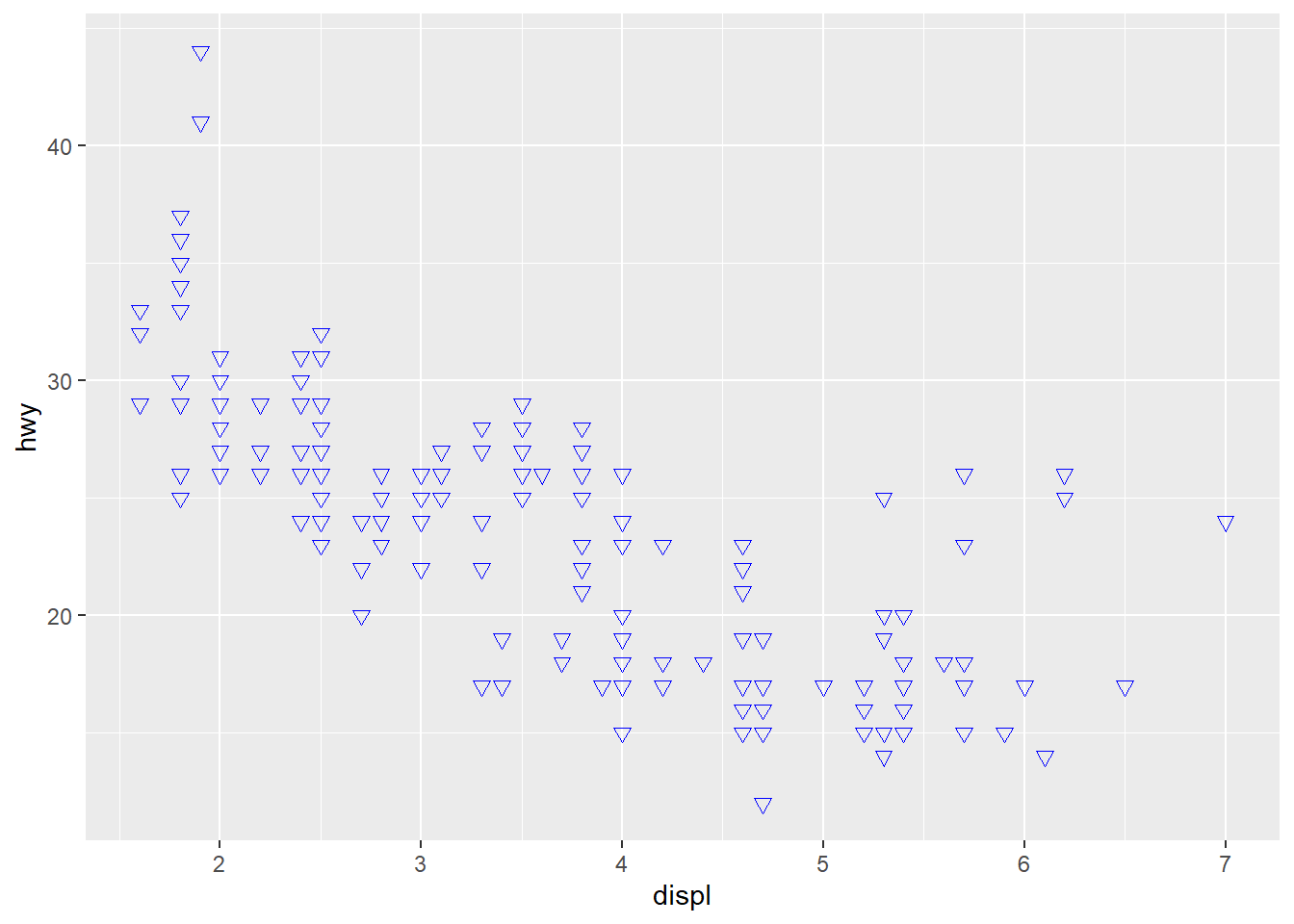
Os pontos não ficam azuis porque você não está especificando cores! Dentro da função aes() você está especificando variáveis para serem mapeadas a uma escala de cores. Portanto, o ggplot interpreta “blue” como uma variável sem nome que tem o valor “blue” e mapeia ela para a escala de cores padrão, que é vermelha. Se você quer controlar apenas a “aparência” dos pontos e não está preocupada em mapear nenhuma variável, você pode passar essa estética fora da função aes().
ggplot(data = mpg) +
geom_point(
mapping = aes(x = displ, y = hwy), # aqui acabam os mapeamentos estéticos
color = "blue", # alteração apenas na aparência do geom
size = 2, # alteração apenas na aparência do geom
shape = 6 # alteração apenas na aparência do geom
)
- Utilizando o banco
mpg, faça o diagrama de dispersão dedisplporhwye mapeie a cor paraclass, o tamanho paracyle a forma paramanufacturer. Como esses atributos estéticos se comportam diferente para variáveis categóricas vs contínuas?
ggplot(mpg, aes(
displ,
hwy,
color = class,
size = cyl,
shape = manufacturer)) +
geom_point()
## Warning: The shape palette can deal with a maximum of 6 discrete values because
## more than 6 becomes difficult to discriminate; you have 15. Consider
## specifying shapes manually if you must have them.
## Warning: Removed 112 rows containing missing values (geom_point).
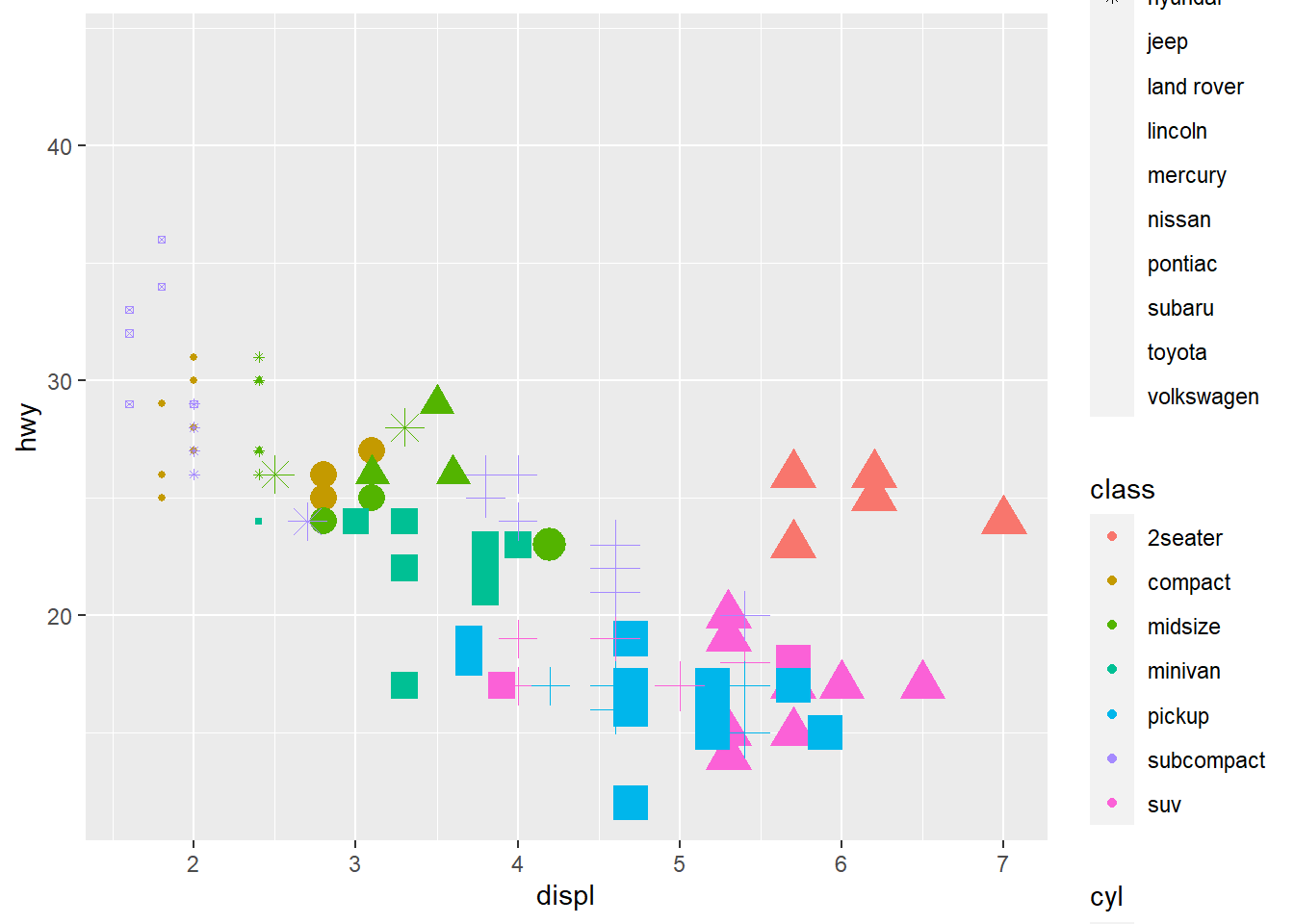
Ao cumprir as instruções como dadas, logo de cara você recebe um aviso do ggplot2. A paleta de “shapes” só recebe por padrão 6 shapes diferentes, porque de acordo com o autor, mais de 6 torna difícil de distinguir. Mas eu sou teimoso.
ggplot(mpg, aes(
displ,
hwy,
color = class,
size = cyl,
shape = manufacturer)) +
geom_point() +
scale_shape_manual(values = 1:15, guide = "legend")
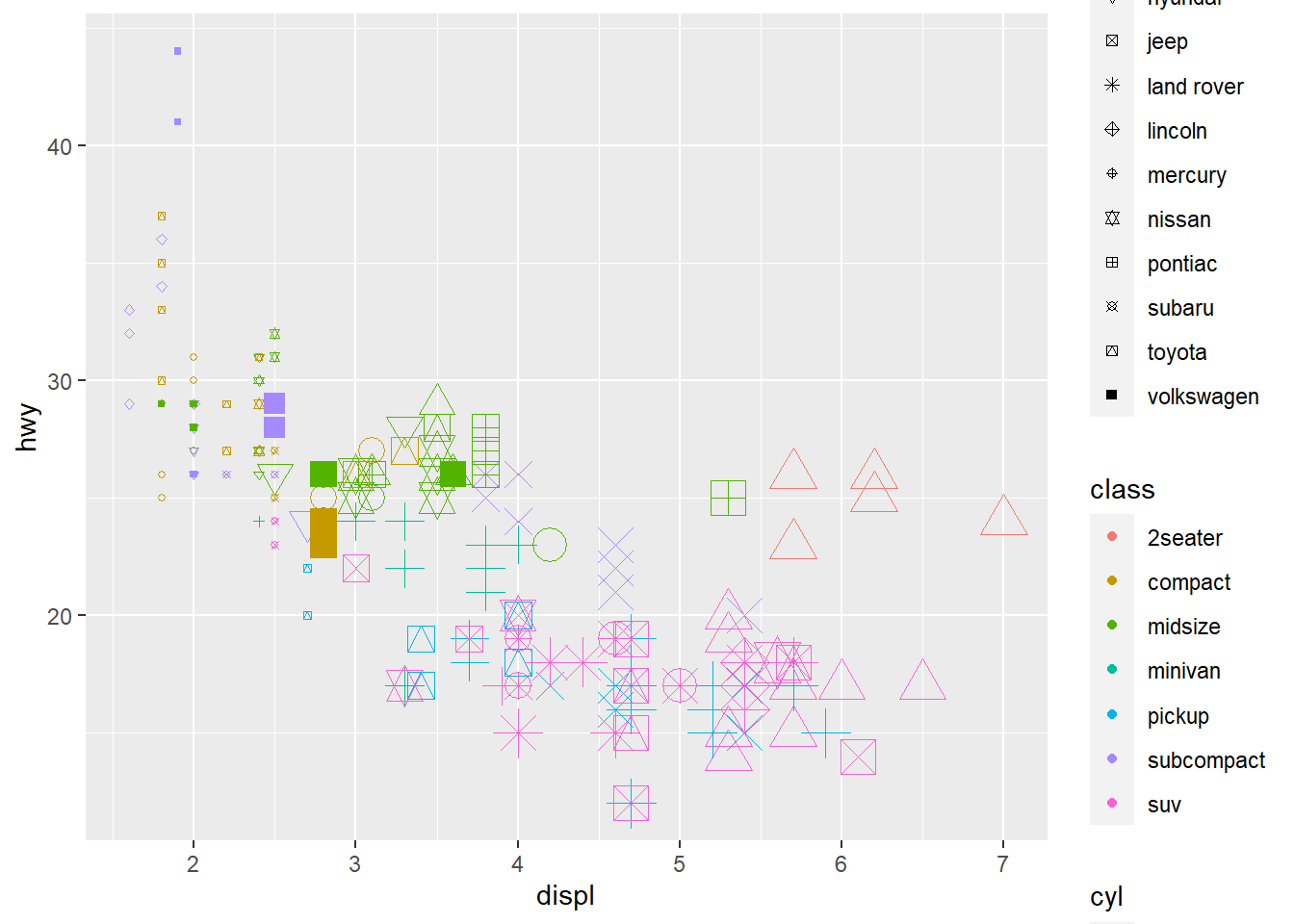
Esse gráfico é mais um exemplo para vocês verem como diferentes escalas se comportam. A variável cyl é numérica e ordenada, então faz sentido colocá-la num mapeamento como size, já que visualmente é possível indicar que a grandeza aumenta com o tamanho. Classe é uma variável categórica, então ela fica melhor em mapeamentos que ressaltam diferenças entre as categorias, como colors ou shapes. O pacote também impõe algumas restrições sobre o que é possível mapear. Por exemplo, ele retorna erro se você tenta mapear uma variável discreta para uma escala contínua.
ggplot(mpg, aes(displ, hwy, color = class)) +
geom_point() +
scale_color_continuous()
## Error: Discrete value supplied to continuous scale

Experimentem tentar mapear diferentes variáveis no banco mpg para as diferentes escalas e vejam os resultados. Em alguns casos, é possível, mas o gráfico é pouco informativo, em outros, você verá mensagens de erro.
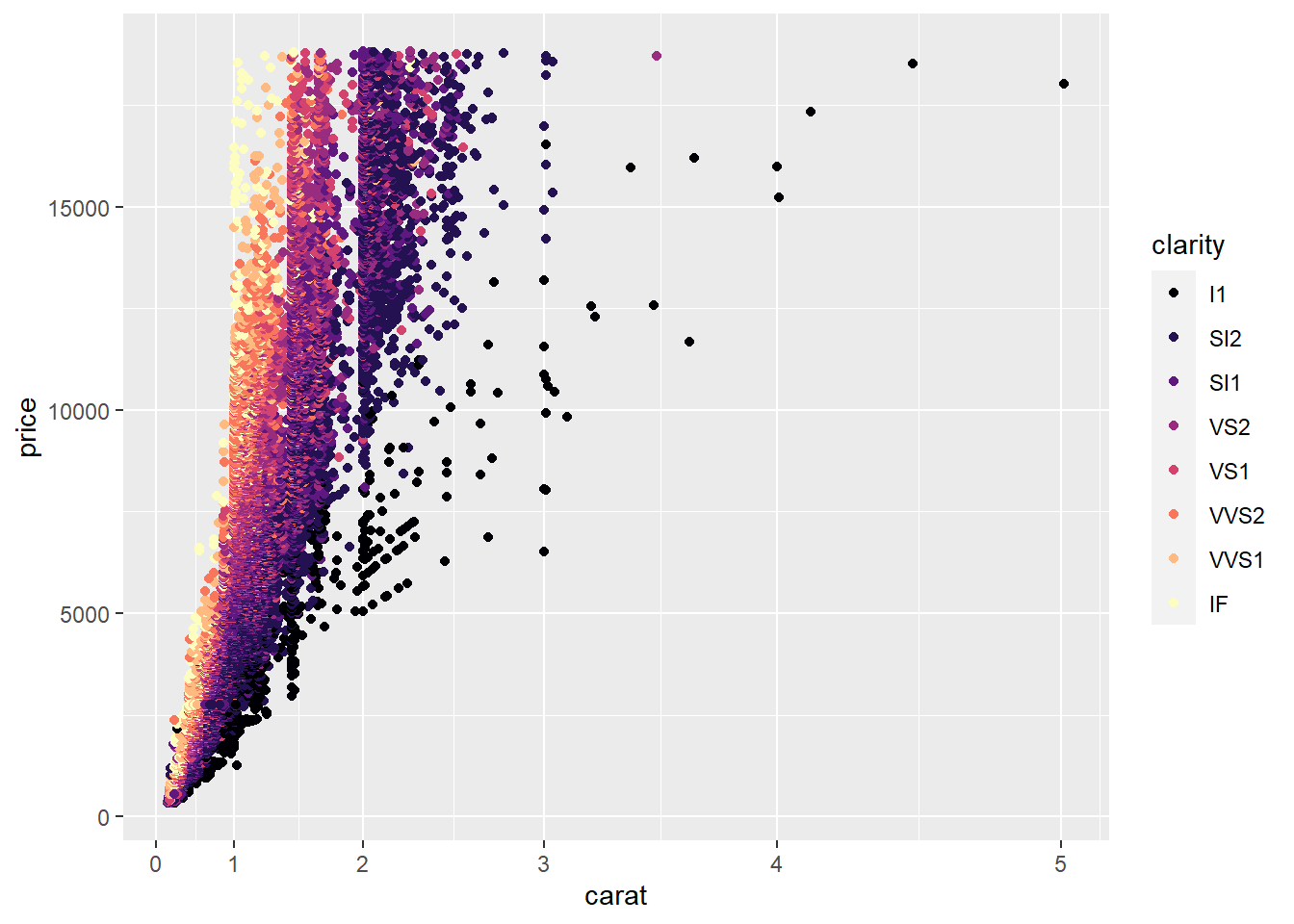
- Utilizando o
diamonds, crie um diagrama de dispersão que relacionecaratcomprice. Explore algumas outras variáveis utilizando escalas de cor para ver se você identifica algum padrão. Aplique transformações nas variáveis que você considerar justificadas.
Esse exercício não tem uma resposta correta. O objetivo era que vocês explorassem as transformações estatísticas e as escalas de cores diferentes presentes no ggplot, através do argumento trans, ou mesmo fazer outras transformações que interessassem vocês nas variáveis. Abaixo um exemplo de transformação de Yeo-Johnson, um tipo de transformação BoxCox que aceita valores negativos e uma das escalas de cor do pacote viridis.
ggplot(diamonds, aes(carat, price, color = clarity)) +
geom_point() +
scale_x_continuous(trans = scales::yj_trans(p = 2)) +
scale_color_viridis_d(option = "magma")
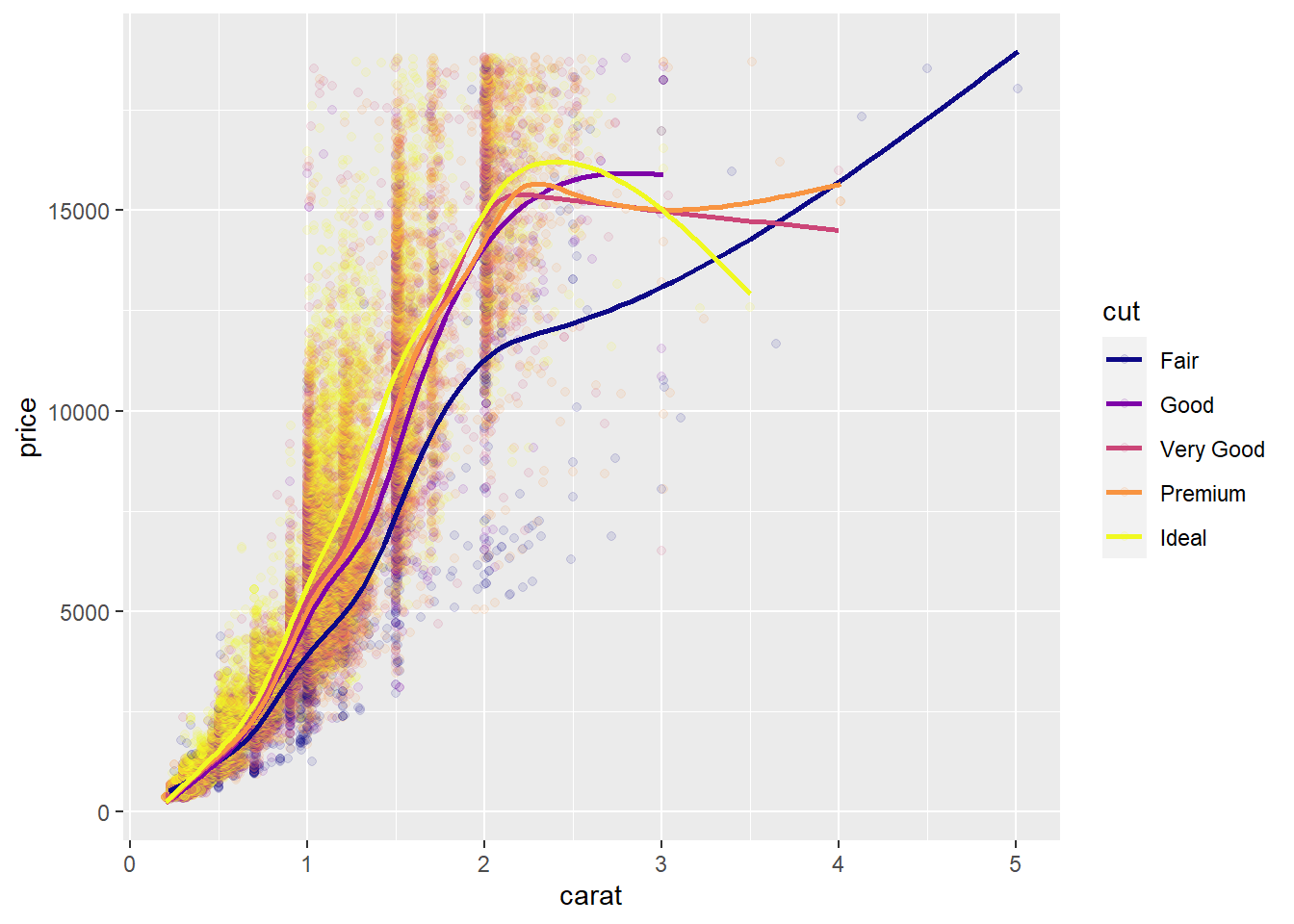
- Ainda continuando o exemplo anterior, aplique um
geom_smoothutilizando várias opções demethodpara as variáveis originais ou transformadas.
Segundo a mesma lógica, o objetivo era explorar as opções de visualização de modelos simples através do argumento method. Abaixo um exemplo de gam. Uma mudança que fiz foi usar a variável cut ao invés de clarity, porque o gráfico não-transformado de clarity estava muito poluído.
ggplot(diamonds, aes(carat, price, color = cut)) +
geom_point(alpha = 0.1) + # pontos translúcidos para reduzir a poluição
geom_smooth(method = "gam", se = FALSE) +
scale_color_viridis_d(option = "plasma")
## `geom_smooth()` using formula 'y ~ s(x, bs = "cs")'
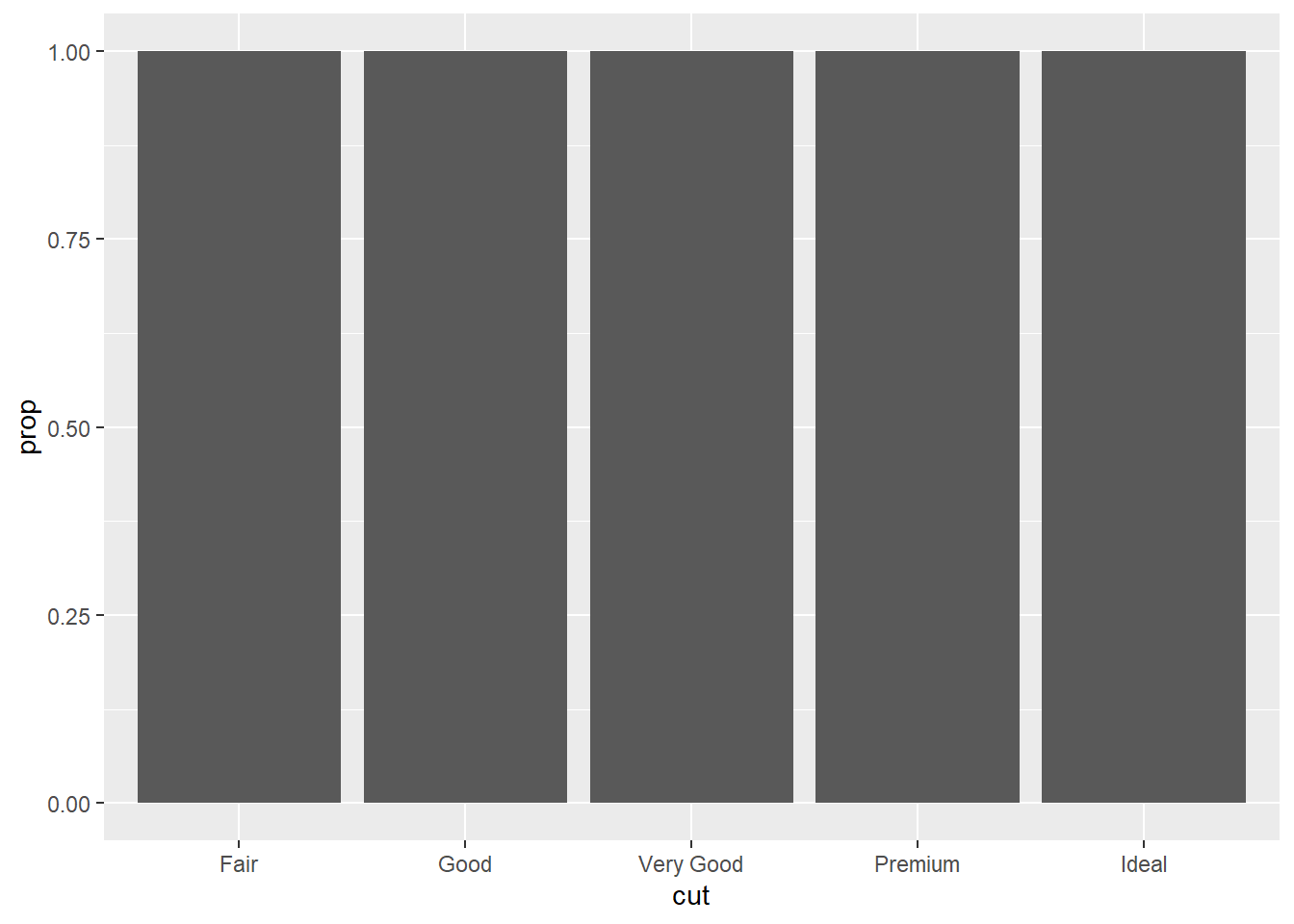
- No nosso gráfico de barras usando
stat(prop)a gente precisou colocargroup = 1, porque? Qual é a diferença entre esses dois códigos?
ggplot(data = diamonds) +
geom_bar(mapping = aes(x = cut, y = after_stat(prop)))
ggplot(data = diamonds) +
geom_bar(mapping = aes(x = cut, fill = color, y = after_stat(prop)))
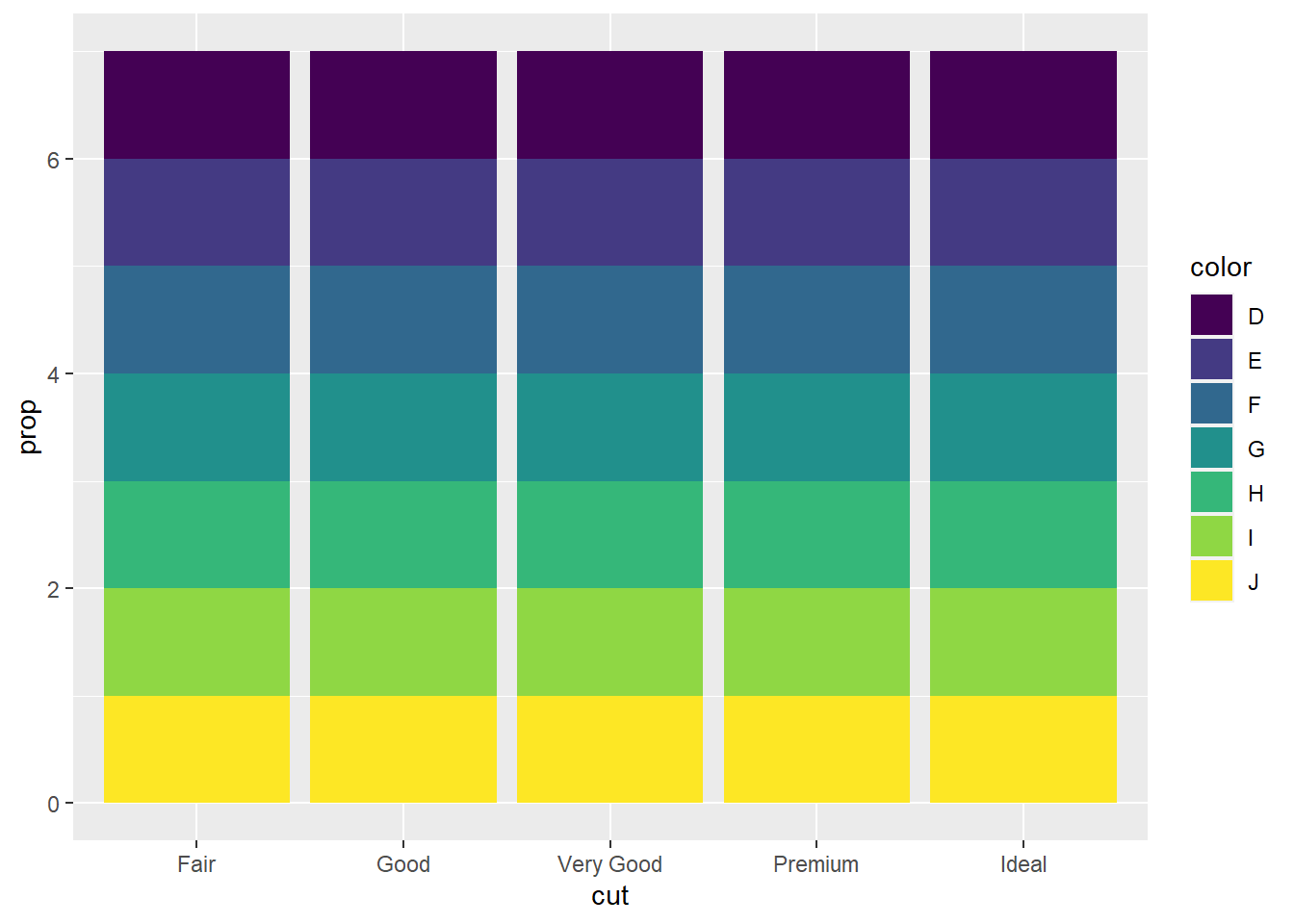
Acabei explicando isso na aula, devido a uma pergunta, mas para quem perdeu, trata-se do comportamento padrão quando há proporções: cada barra terá sua própria proporção e todas somarão a 100%. O uso de group = 1 indica à função que as proporções que somam a 100% são o total dos níveis do fator e não cada nível individualmente.
ggplot(data = diamonds) +
geom_bar(mapping = aes(x = cut, y = after_stat(prop), group = 1))
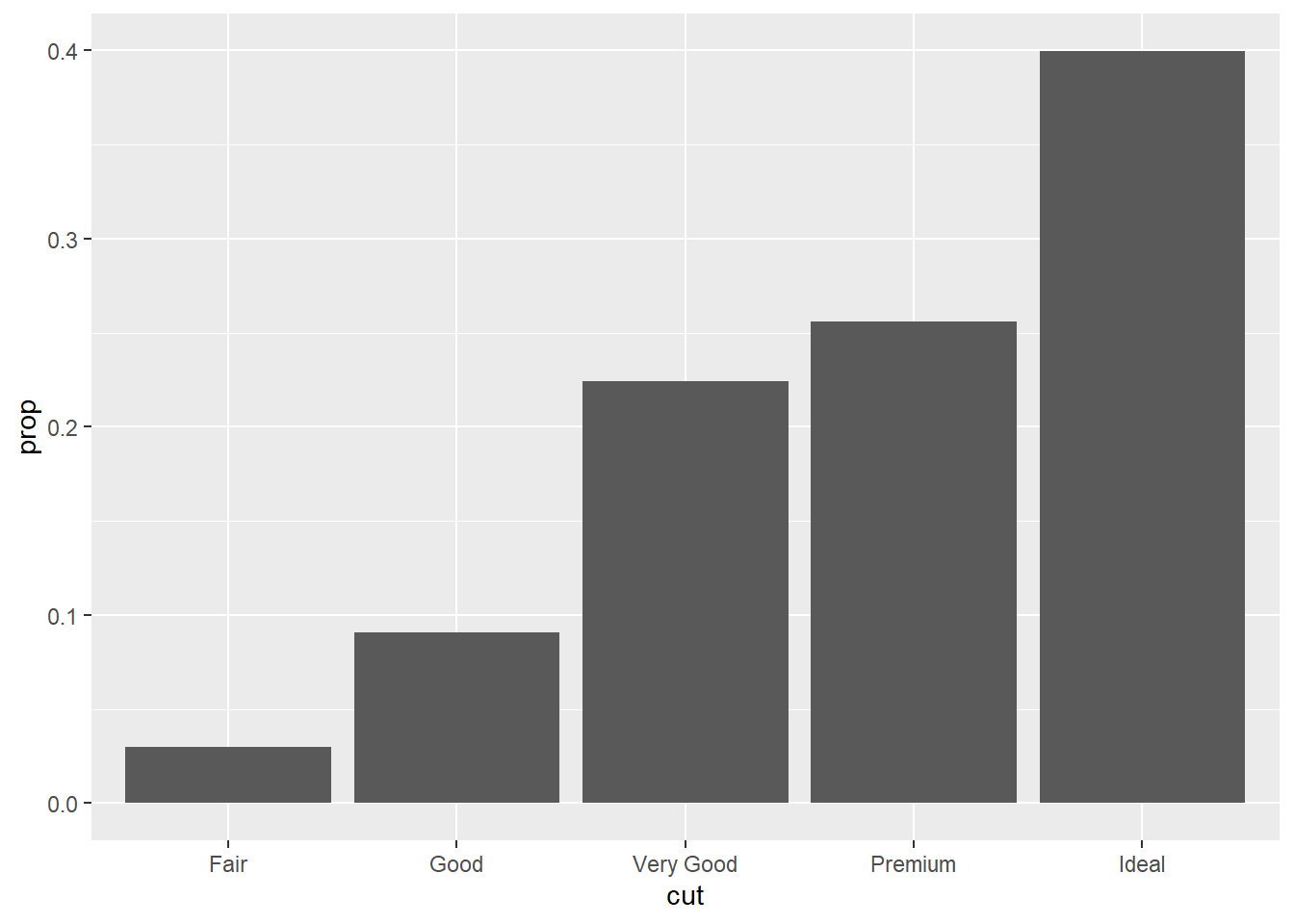
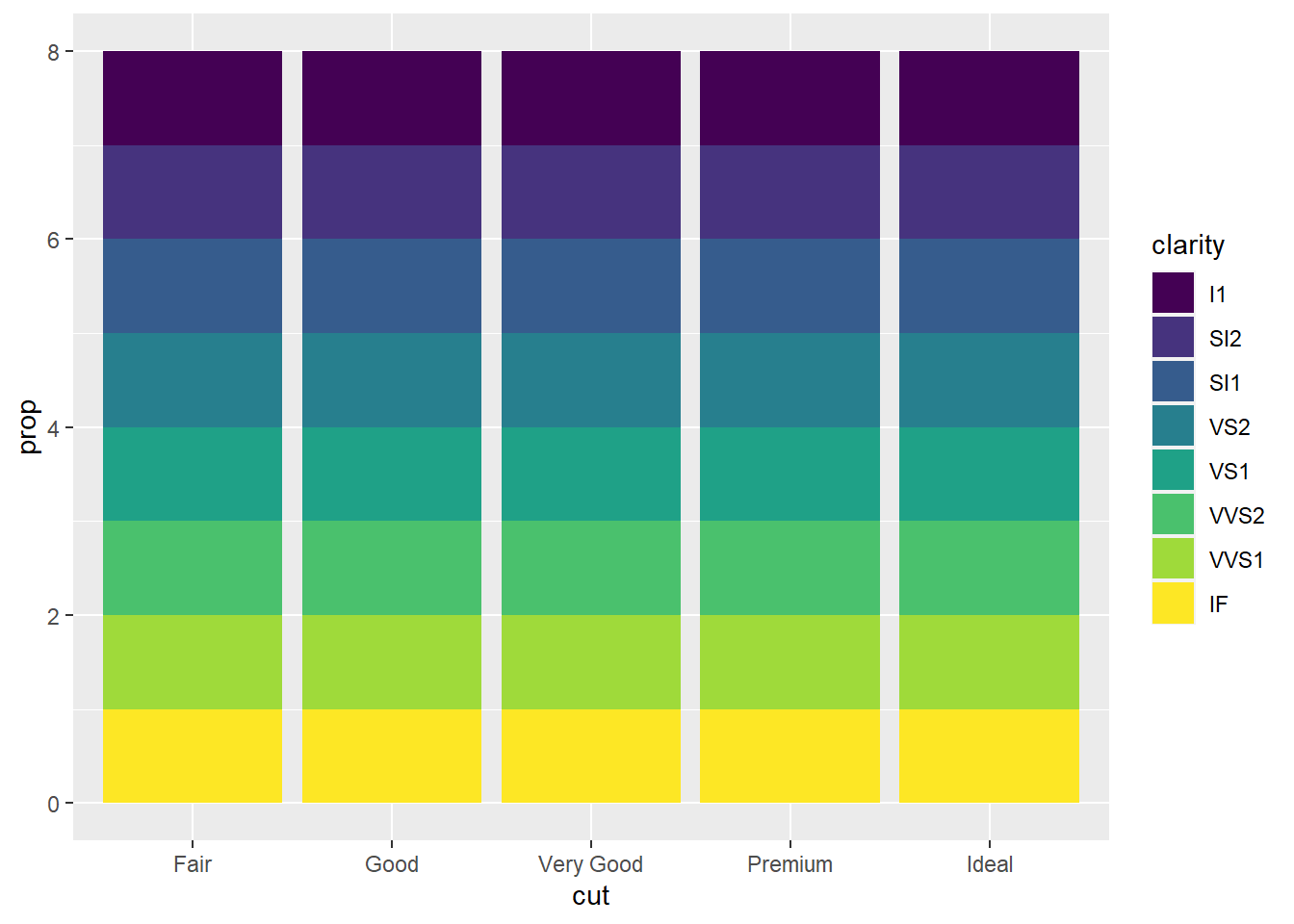
# No caso em que há um "fill", precisamos normalizar as alturas das barras
ggplot(data = diamonds) +
geom_bar(mapping = aes(
x = cut,
y = stat(prop),
fill = clarity
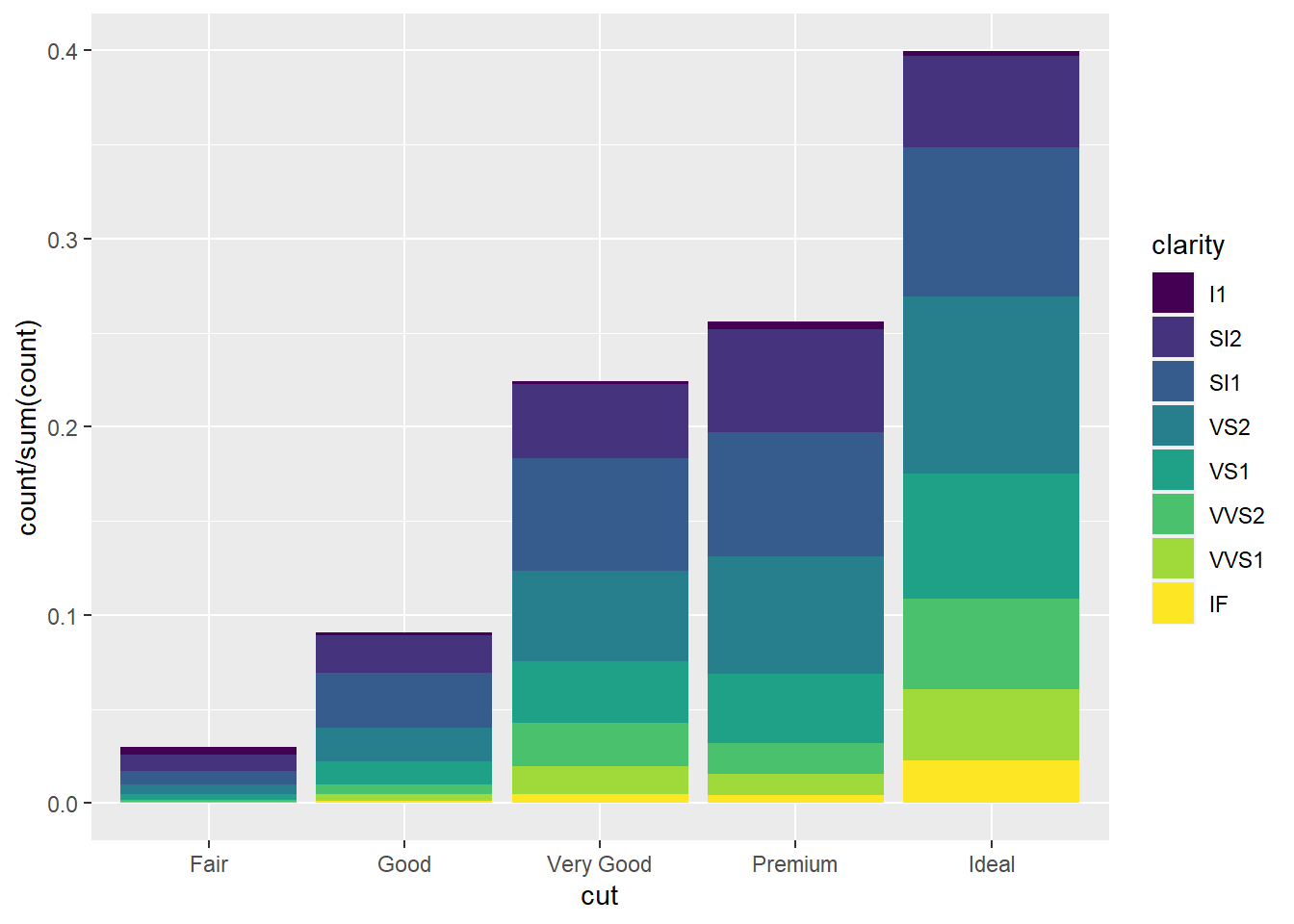
))
ggplot(data = diamonds) +
geom_bar(mapping = aes(
x = cut,
y = stat(count)/sum(stat(count)),
fill = clarity
))
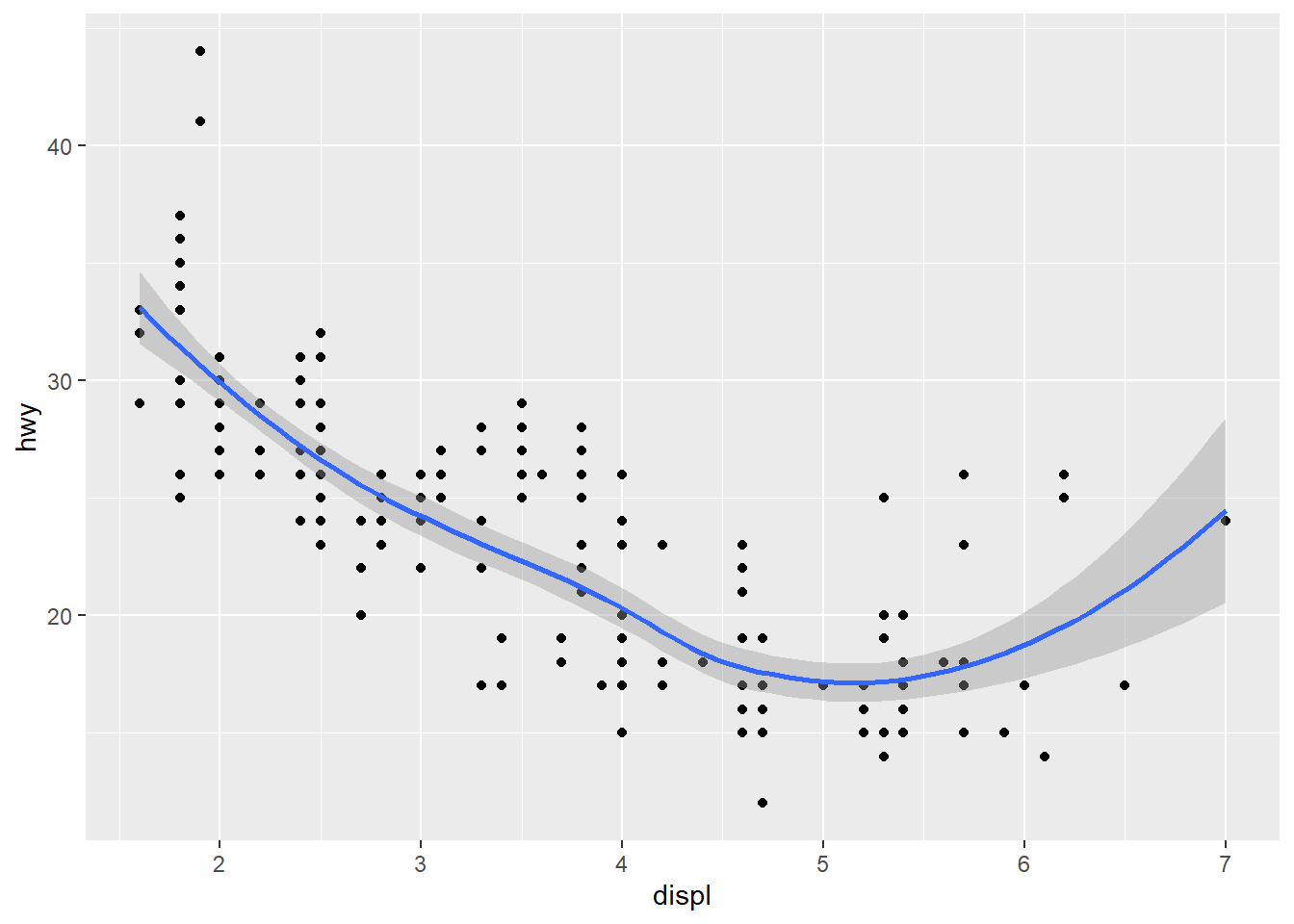
stat_smoothé muito parecido comgeom_smooth, mas há uma diferença sutil. Compare os códigos abaixo.
geom_smooth chama stat_smooth quando você utiliza a função para calcular as “médias condicionais” que correspondem a linha de tendência desenhada no gráfico. É assim com todos os geoms no pacote. Há uma conexão entre o objeto geométrico e uma transformação estatística. Mesmo que seja a transformação _identity, que mantém a variável exatamente como ela estava no dado. A grande vantagem de construir um gráfico com stat_smooth ao invés de geom_smooth é que você pode especificar outro objeto geométrico que não seja o padrão (geom_line + geom_ribbon). É isso que os gráficos abaixo demonstram.
ggplot(mpg, aes(displ, hwy)) +
geom_point() +
geom_smooth()
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
ggplot(mpg, aes(displ, hwy)) +
geom_point() +
stat_smooth(geom = "step")
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
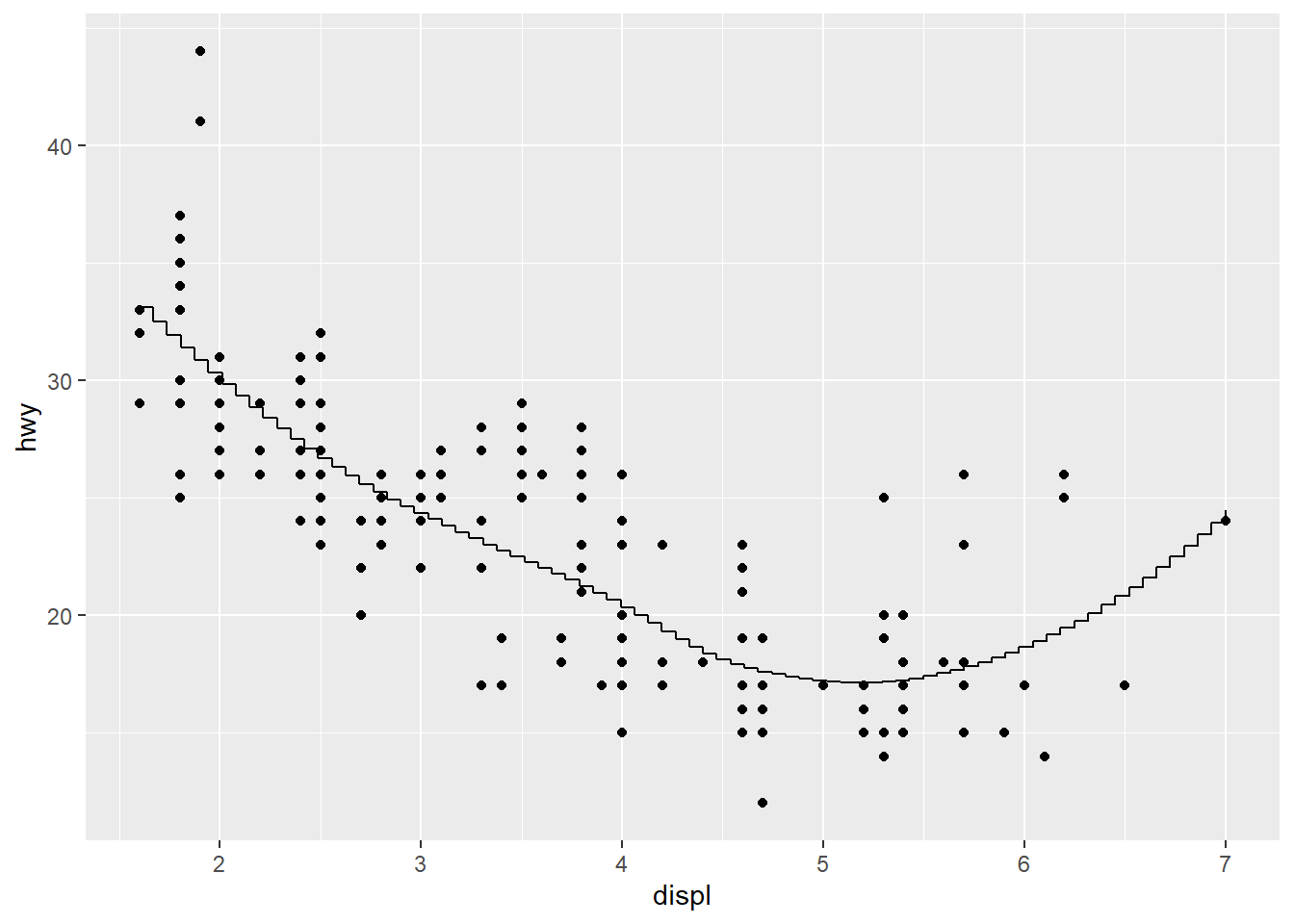
ggplot(mpg, aes(displ, hwy)) +
geom_point() +
stat_smooth(geom = "linerange")
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
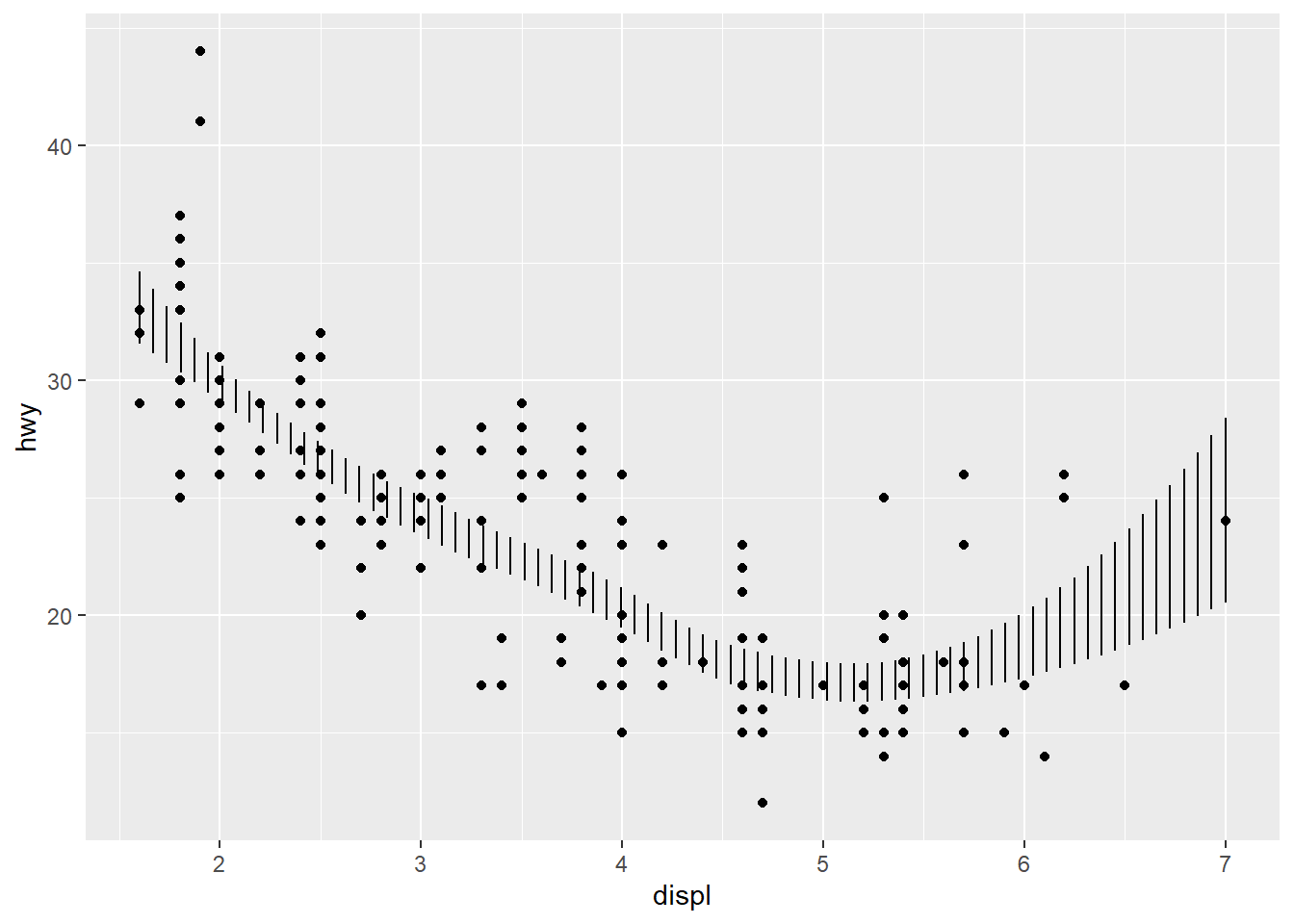
ggplot(mpg, aes(displ, hwy)) +
geom_point() +
stat_smooth(geom = "errorbar")
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
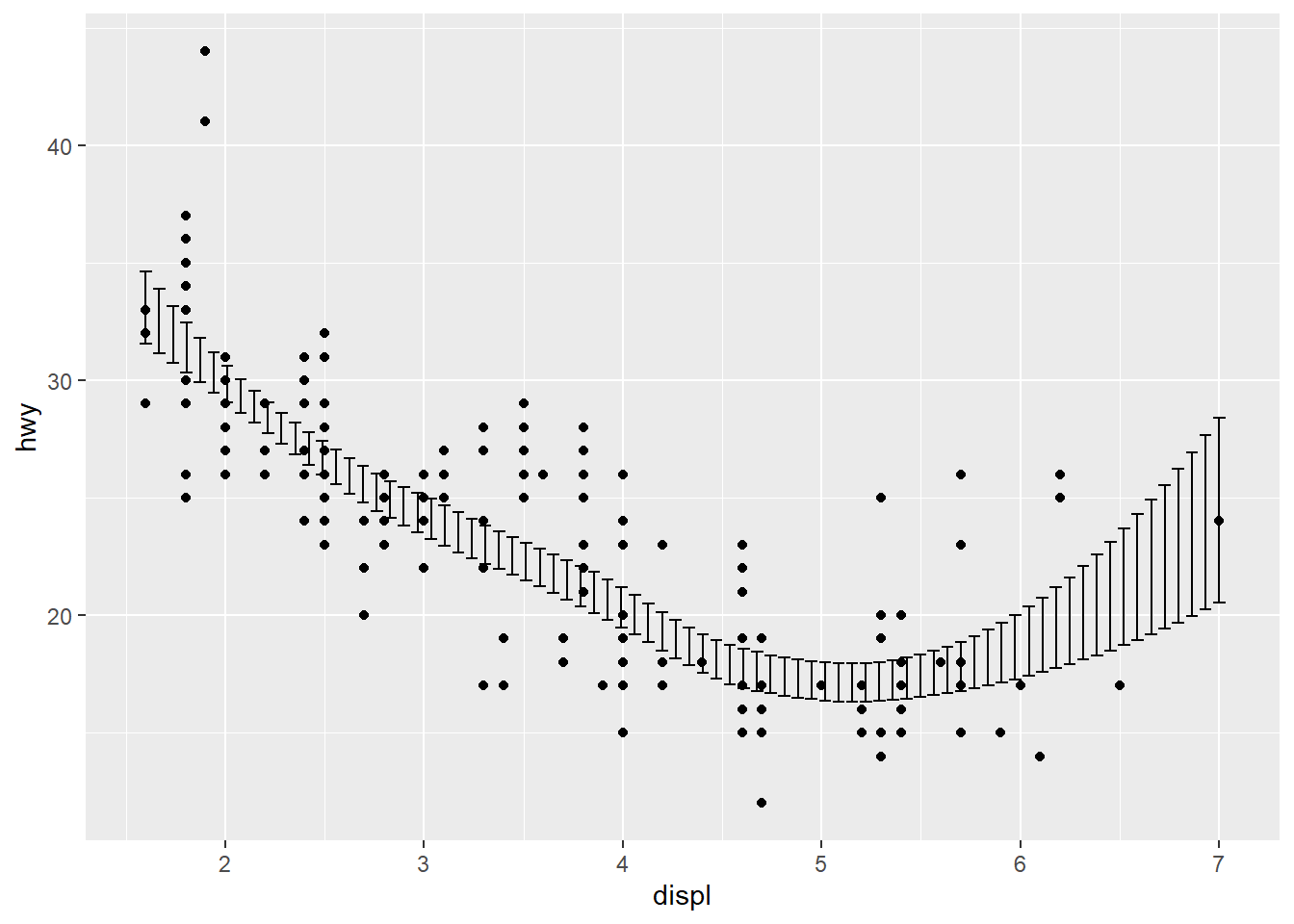
ggplot(mpg, aes(displ, hwy)) +
geom_point() +
stat_smooth(geom = "crossbar")
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
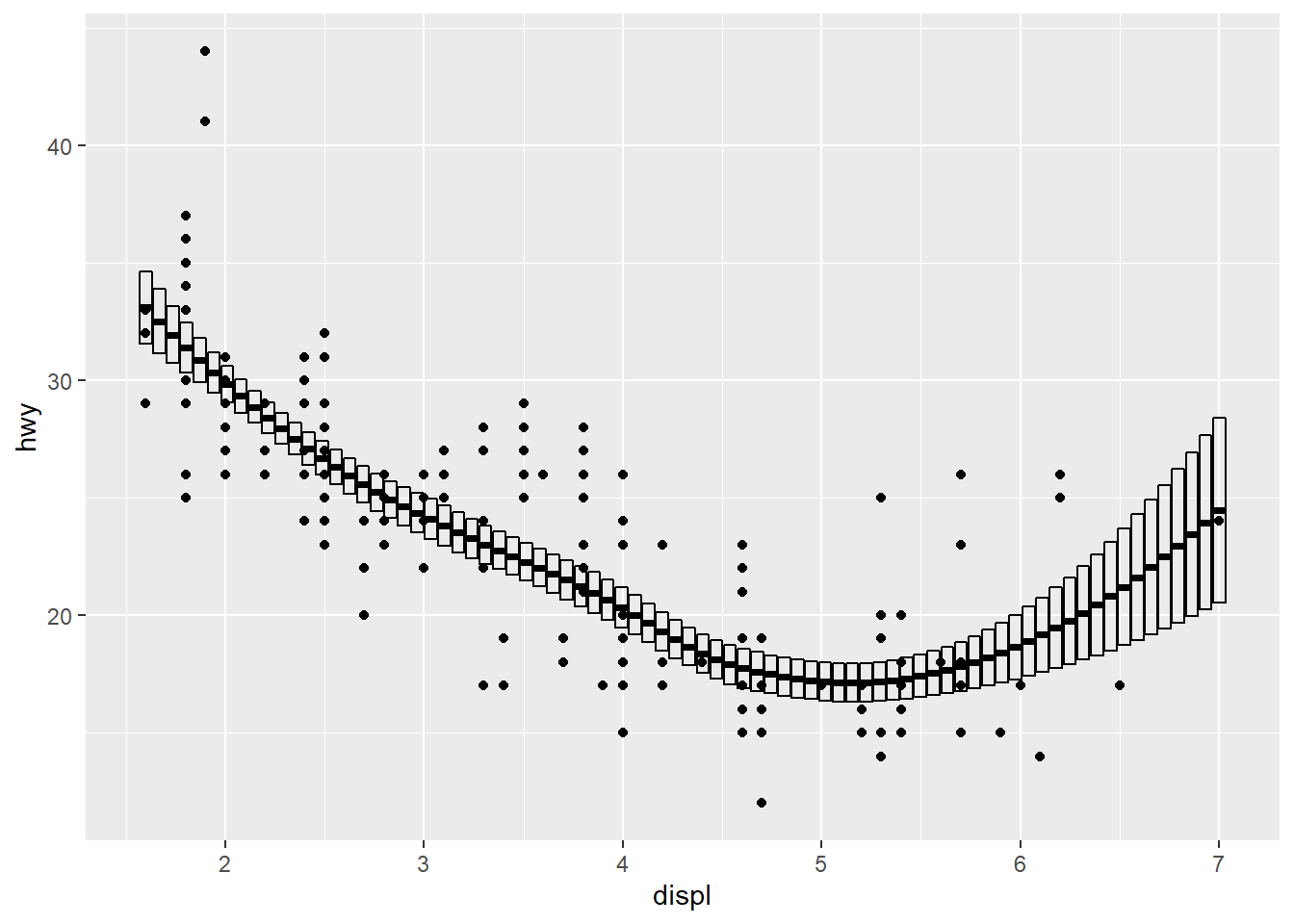
- Usando o
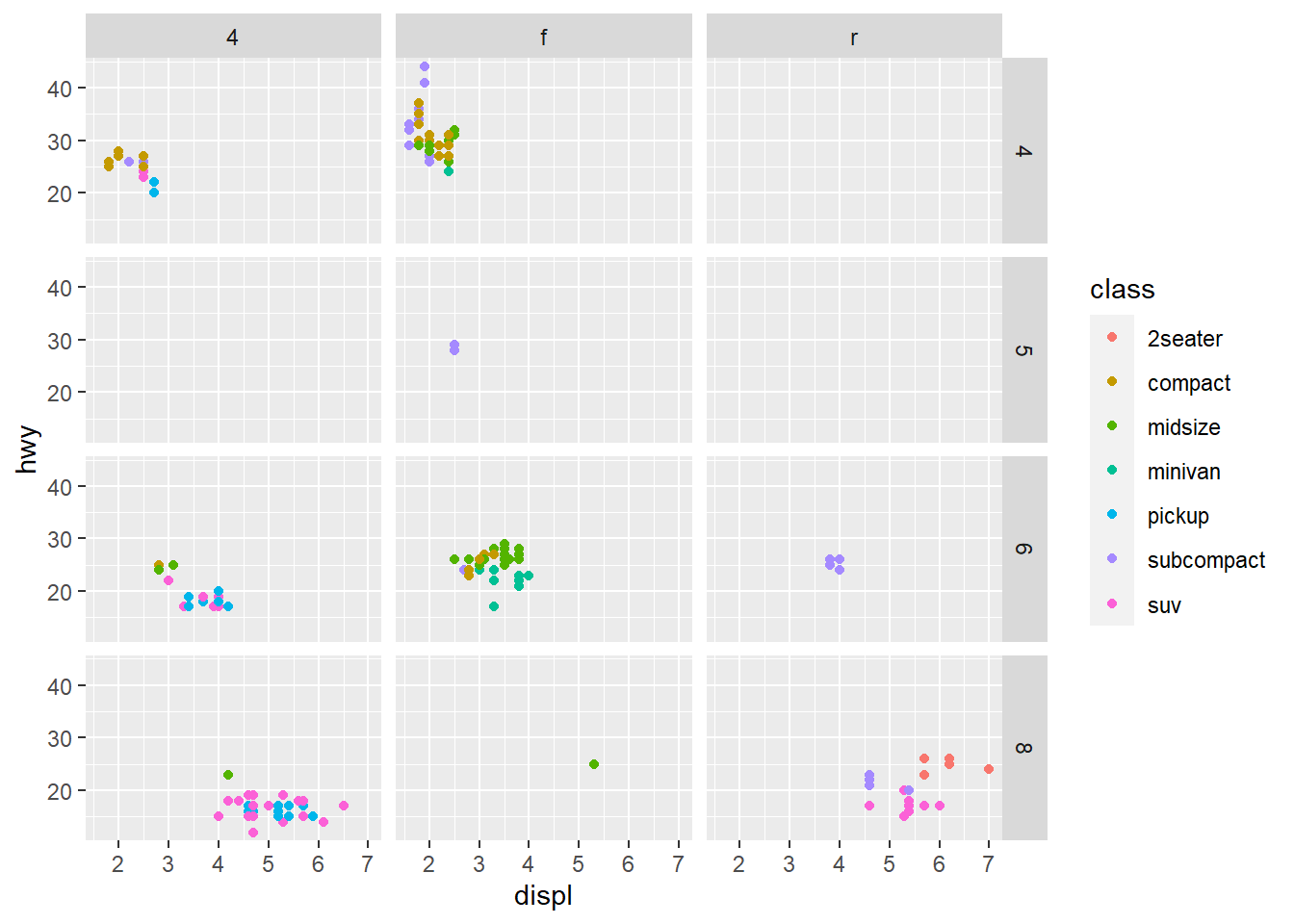
mpgefacet_grid, crie um scatterplot que contenhadisplno eixo x,hwyno eixo y,classna cor,drvnas facetas-coluna ecylnas facetas linha.
Esse aqui é para demonstrar o uso de facet_grid, que permite especificar fatores de classificação diferentes nas linhas e colunas, diferente de facet_wrap mostrado na aula, que só permite especificar uma dimensão.
ggplot(mpg, aes(x = displ, y = hwy, color = class)) +
geom_point() +
facet_grid(cyl ~ drv)
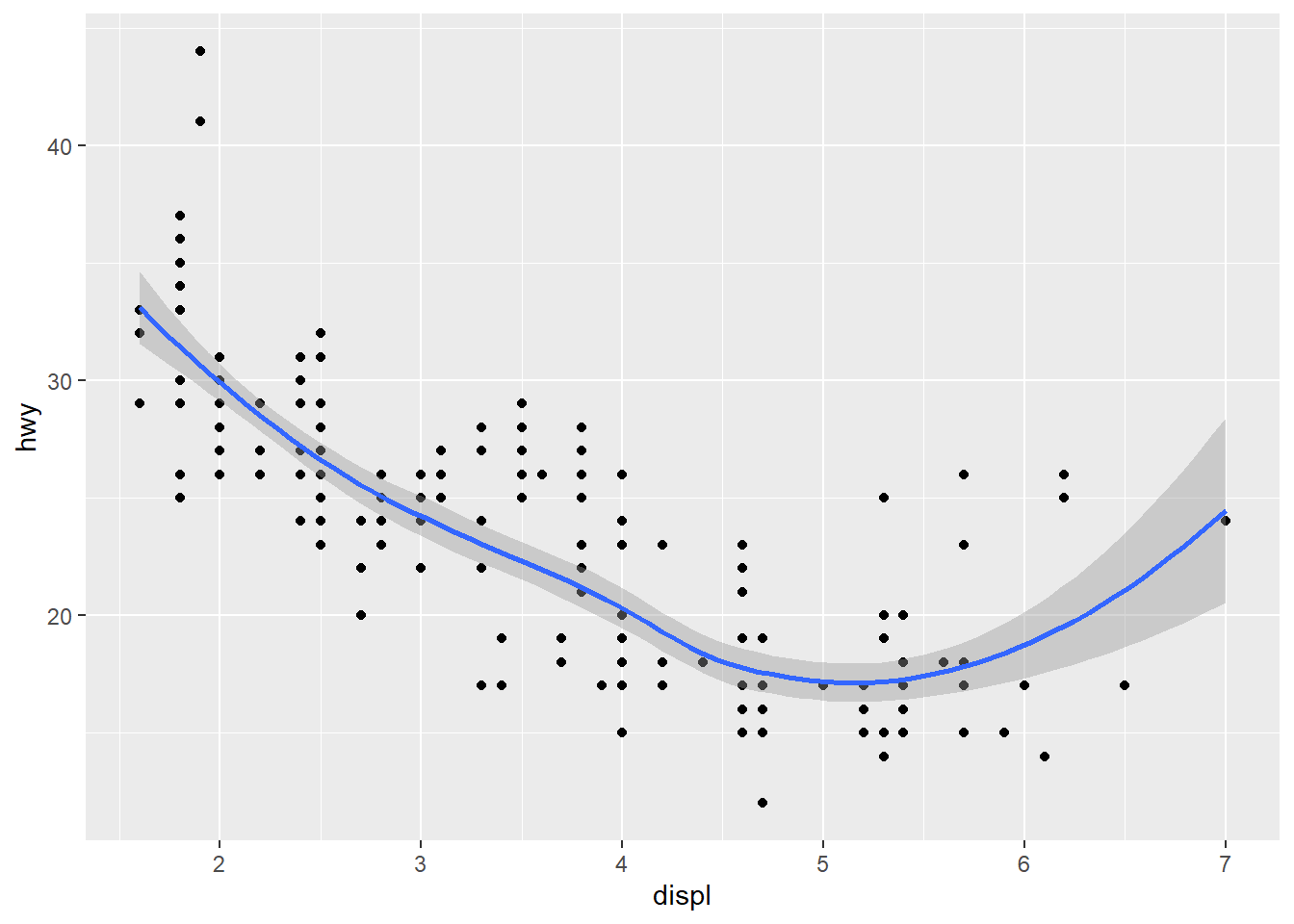
- Você acha que os dois gráficos abaixo ficarão diferentes um do outro? Porque? Tente responder antes de rodar o código.
ggplot(data = mpg, mapping = aes(x = displ, y = hwy)) +
geom_point() +
geom_smooth()
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
ggplot() +
geom_point(data = mpg, mapping = aes(x = displ, y = hwy)) +
geom_smooth(data = mpg, mapping = aes(x = displ, y = hwy))
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
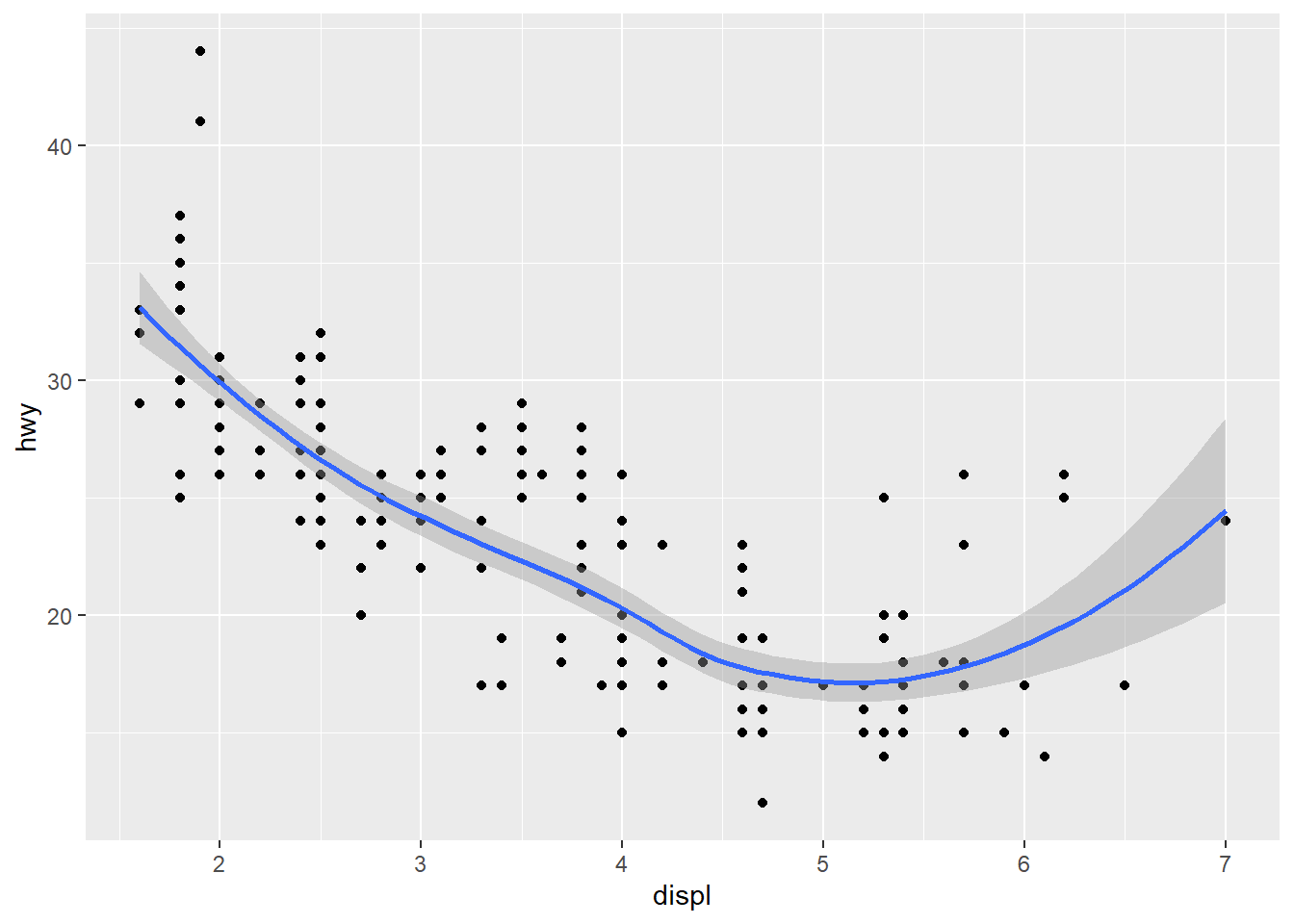
Mesmo antes de rodar o código, o observador astuto notará que os mapeamentos locais no segundo gráfico são idênticos entre si e aos mapeamentos globais, então os dois gráficos são iguais.
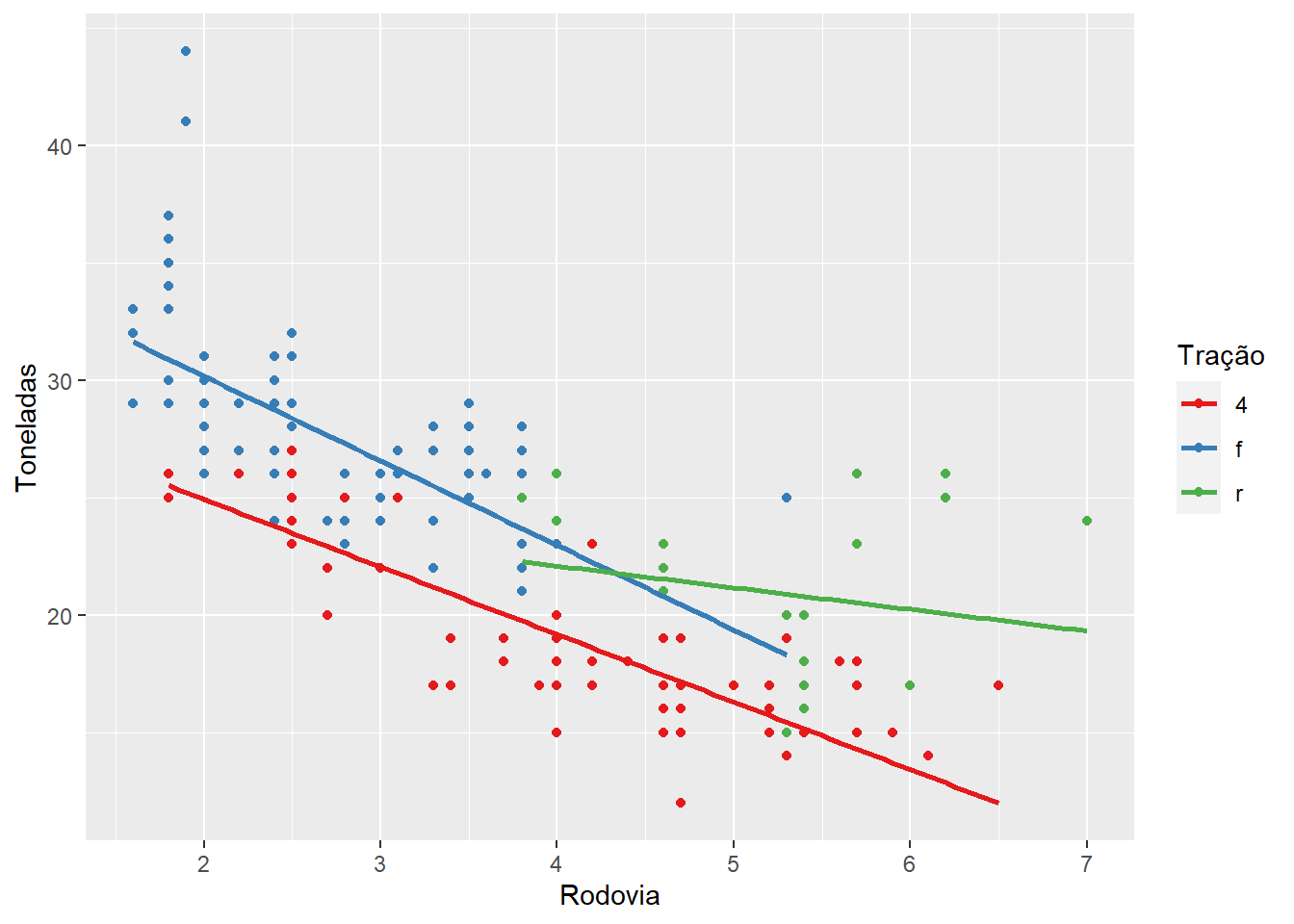
- Tente recriar o seguinte gráfico
O objetivo dessa era fazer vocês fuçarem um pouco na ajuda para tentar recriar o mais fielmente possível o gráfico final. Não precisava ter acertado, o objetivo era chegar o mais próximo possível.
ggplot(mpg, aes(displ, hwy, color = drv)) +
geom_point() +
geom_smooth(method = lm, se = FALSE) +
labs(x = "Rodovia", y = "Toneladas", color = "Tração") +
scale_color_brewer(palette = "Set1")
## `geom_smooth()` using formula 'y ~ x'
- Transforme o gráfico seguir em um gráfico de pizza usando
coord_polar.
ggplot(diamonds, aes(cut, fill = cut)) +
geom_bar()
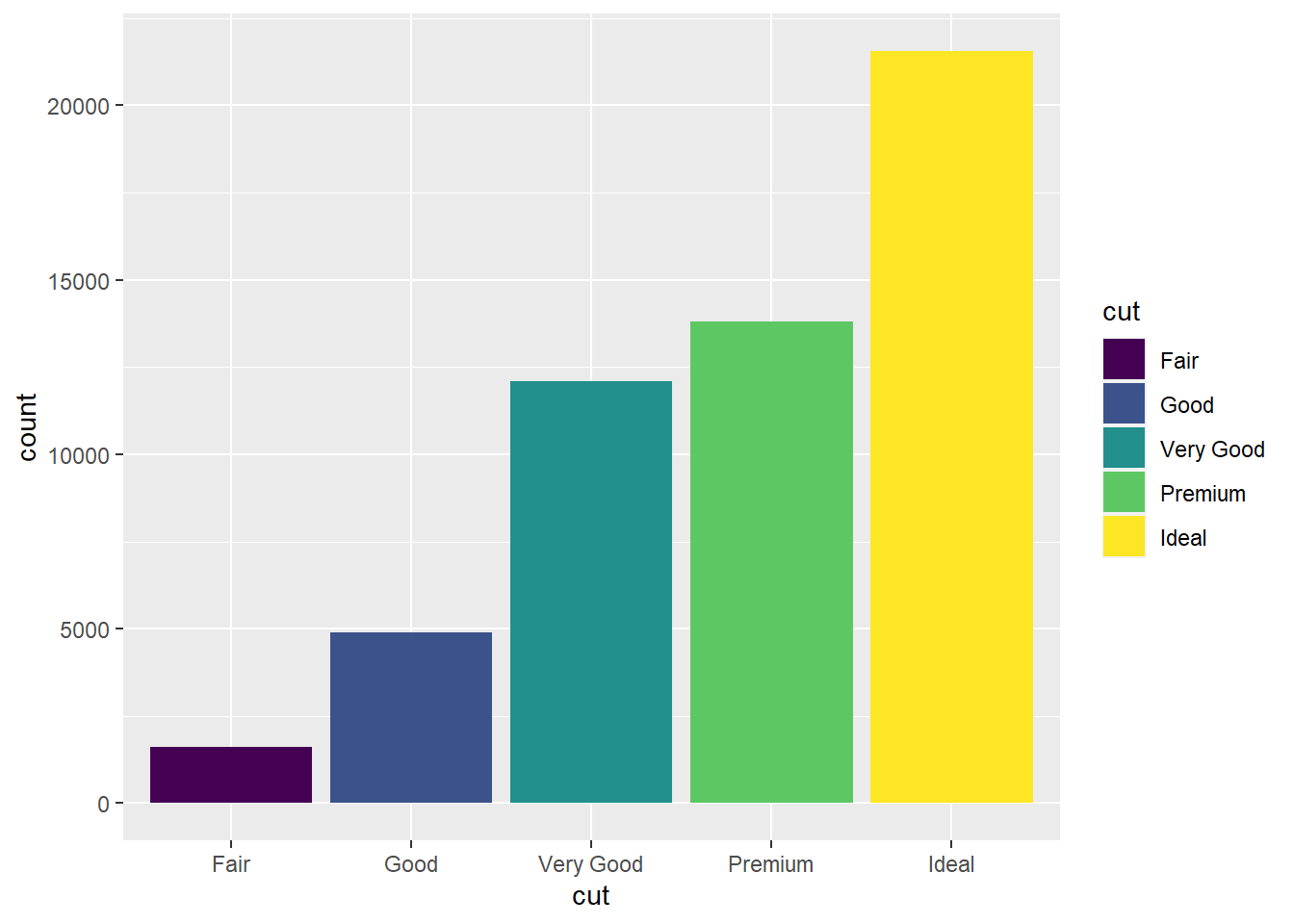
Depois de simplesmente especificar coord_polar, em geral o gráfico fica meio estranho, não tem aquela cara bonita de pizza. É preciso corrigir os seguintes problemas:
- A largura das barras deve ser igual a proporção das contagens, mas a altura deve ser igual a 1! Portanto, eu inverto as coisas e passo as contagens/proporções para “x” e “y” fica com o valor fixo = 1.
ggplot(diamonds,
aes(
# calculando as proporções do total,
# também funciona com o padrão stat(count)
x = stat(count)/sum(stat(count)),
y = 1, # altura igual a 1
fill = cut)) + # cores
geom_bar() +
coord_polar() + # coordenadas polares
# opcional: remover aspectos do tema para um visual mais clean
theme_void()
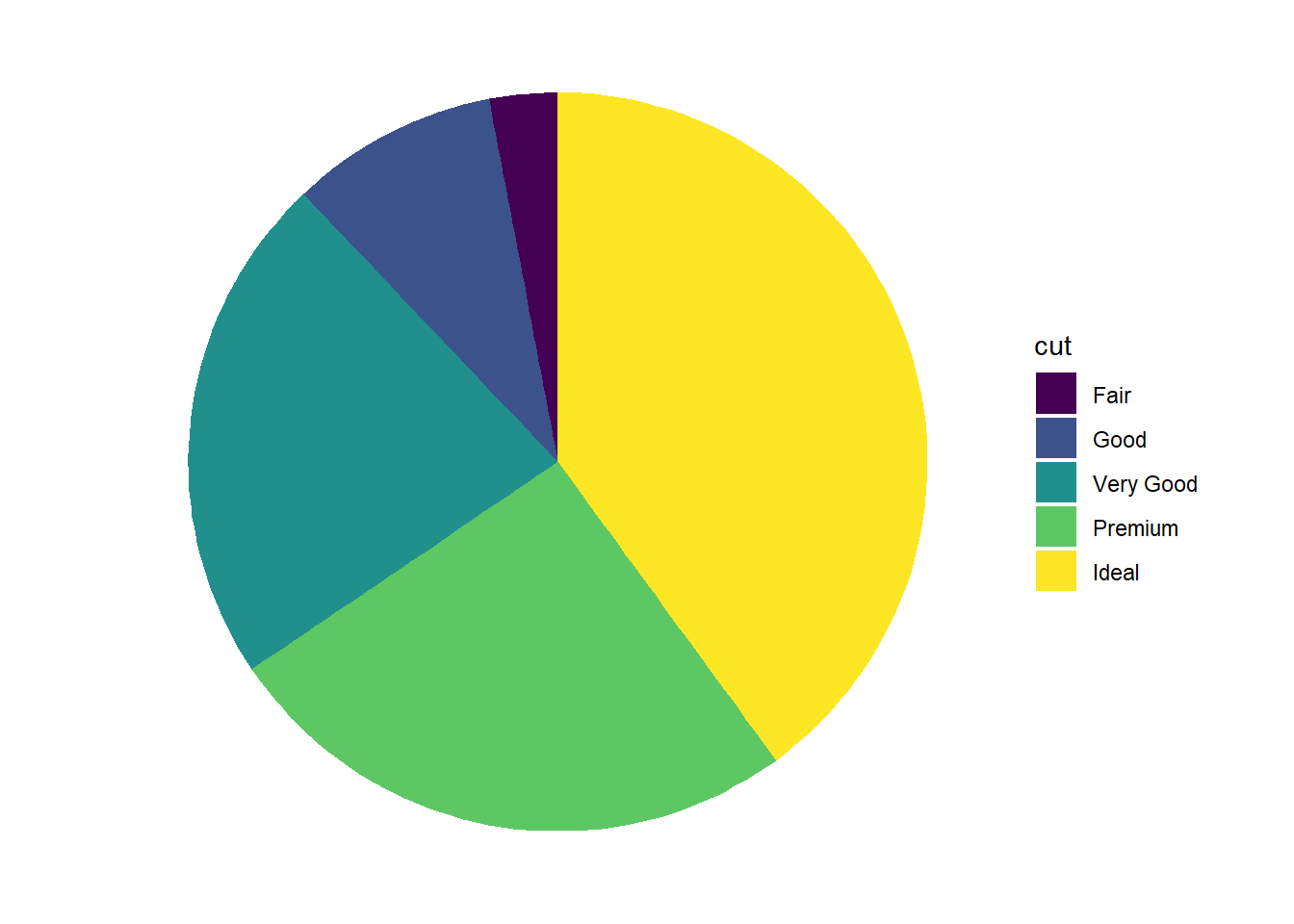
Como desafio, tentem adicionar elementos textuais das proporções no gráfico. O problema a ser resolvido é como posicionar o texto num sistema de coordenadas polares. Boa sorte!
Gráficos de pizza são polêmicos na análise de dados porque nossos olhos não captam bem diferenças entre formatos curvos e complexos, então a comparação entre as categorias fica prejudicada se houverem mais de 2 ou 3. Eu sempre dou preferência para barras. Há um tipo de gráfico de pizza melhorzinho chamado “donut plot”, em que o meio do círculo é oco, mas eu ainda prefiro as barras.
purrr
- Utilize uma das funções
map_para:
Nos exemplos abaixo, eu utilizo sempre str() no final para facilitar a visualização das listas, mas não é obrigatório utilizar esse comando.
1. Calcular a média de cada coluna em `mtcars`.
map(mtcars, mean) %>% str()
## List of 11
## $ mpg : num 20.1
## $ cyl : num 6.19
## $ disp: num 231
## $ hp : num 147
## $ drat: num 3.6
## $ wt : num 3.22
## $ qsec: num 17.8
## $ vs : num 0.438
## $ am : num 0.406
## $ gear: num 3.69
## $ carb: num 2.81
2. Determinar o tipo de cada coluna em `flights`.
flights %>% map(class) %>% str()
## List of 19
## $ year : chr "integer"
## $ month : chr "integer"
## $ day : chr "integer"
## $ dep_time : chr "integer"
## $ sched_dep_time: chr "integer"
## $ dep_delay : chr "numeric"
## $ arr_time : chr "integer"
## $ sched_arr_time: chr "integer"
## $ arr_delay : chr "numeric"
## $ carrier : chr "character"
## $ flight : chr "integer"
## $ tailnum : chr "character"
## $ origin : chr "character"
## $ dest : chr "character"
## $ air_time : chr "numeric"
## $ distance : chr "numeric"
## $ hour : chr "numeric"
## $ minute : chr "numeric"
## $ time_hour : chr [1:2] "POSIXct" "POSIXt"
3. Computar o número de valores únicos em cada coluna de `iris`.
flights %>% map(unique) %>% map(length) %>% str()
## List of 19
## $ year : int 1
## $ month : int 12
## $ day : int 31
## $ dep_time : int 1319
## $ sched_dep_time: int 1021
## $ dep_delay : int 528
## $ arr_time : int 1412
## $ sched_arr_time: int 1163
## $ arr_delay : int 578
## $ carrier : int 16
## $ flight : int 3844
## $ tailnum : int 4044
## $ origin : int 3
## $ dest : int 105
## $ air_time : int 510
## $ distance : int 214
## $ hour : int 20
## $ minute : int 60
## $ time_hour : int 6936
# Sugestão do Pedro Gomes
flights %>% map(n_distinct) %>% str()
## List of 19
## $ year : int 1
## $ month : int 12
## $ day : int 31
## $ dep_time : int 1319
## $ sched_dep_time: int 1021
## $ dep_delay : int 528
## $ arr_time : int 1412
## $ sched_arr_time: int 1163
## $ arr_delay : int 578
## $ carrier : int 16
## $ flight : int 3844
## $ tailnum : int 4044
## $ origin : int 3
## $ dest : int 105
## $ air_time : int 510
## $ distance : int 214
## $ hour : int 20
## $ minute : int 60
## $ time_hour : int 6936
4. Gere 10 distribuições aleatórias (`rnorm`) com médias -10, 0, 10 e 100.
x <- rep(100, 40)
medias <- rep(c(-10, 0, 10, 100), each = 10)
map2(x, medias, ~rnorm(.x, mean = .y)) %>% str()
## List of 40
## $ : num [1:100] -9.96 -9.53 -9.12 -9.64 -10.36 ...
## $ : num [1:100] -11.71 -10.76 -9.41 -9.22 -10.53 ...
## $ : num [1:100] -11.33 -9.51 -8.98 -10.91 -9.52 ...
## $ : num [1:100] -11.4 -10.9 -10.7 -12.1 -9.8 ...
## $ : num [1:100] -11.06 -10.66 -10.33 -9.13 -9.13 ...
## $ : num [1:100] -10.22 -9.59 -9.81 -10.71 -10.24 ...
## $ : num [1:100] -11 -9.14 -10.91 -9.38 -9.63 ...
## $ : num [1:100] -9.83 -9.61 -11.23 -9.68 -10.47 ...
## $ : num [1:100] -9.11 -10.03 -10.62 -9.65 -10.2 ...
## $ : num [1:100] -9.06 -10.41 -10.84 -9.16 -9.41 ...
## $ : num [1:100] -0.8378 0.1102 0.7519 -0.2477 0.0342 ...
## $ : num [1:100] 0.607 -0.902 -2.185 -0.397 -1.019 ...
## $ : num [1:100] -0.452 2.067 1.45 -1.184 0.893 ...
## $ : num [1:100] -0.604 -0.783 1.767 -0.632 1.322 ...
## $ : num [1:100] -2.515 0.43 0.694 1.239 1.091 ...
## $ : num [1:100] -1.462 1.177 -0.376 0.037 0.387 ...
## $ : num [1:100] -0.0803 -0.0421 2.4765 0.0509 -0.1479 ...
## $ : num [1:100] 1.118 0.662 -1.439 -1.074 0.493 ...
## $ : num [1:100] -0.3136 0.4455 -0.947 2.0658 -0.0188 ...
## $ : num [1:100] 0.193 0.625 2.281 0.297 -0.629 ...
## $ : num [1:100] 9.29 9.1 9.54 9.99 10.2 ...
## $ : num [1:100] 7.96 7.32 10.7 9.13 10.3 ...
## $ : num [1:100] 9.96 10.24 10.31 10.54 11.65 ...
## $ : num [1:100] 10.88 10.41 8.19 10.37 10.28 ...
## $ : num [1:100] 11.27 9.74 11.75 11.7 9.31 ...
## $ : num [1:100] 9.62 11.35 10.78 10.79 9.7 ...
## $ : num [1:100] 9.04 10.24 11.85 9.82 10.66 ...
## $ : num [1:100] 10.48 6.97 8.97 9.69 11.03 ...
## $ : num [1:100] 8.48 10.12 9.09 8.18 10.11 ...
## $ : num [1:100] 9.97 9.27 10.19 10.93 11.07 ...
## $ : num [1:100] 100 99.2 99.6 100.3 99.2 ...
## $ : num [1:100] 99 99.9 100.7 98.7 99.7 ...
## $ : num [1:100] 99.6 99.2 100.1 100 99.9 ...
## $ : num [1:100] 100.5 100.4 100.2 98.4 98.9 ...
## $ : num [1:100] 99.8 99.8 100 99.1 101.1 ...
## $ : num [1:100] 100.7 100 100.1 98.6 100 ...
## $ : num [1:100] 99 99.4 100.5 100.1 99.3 ...
## $ : num [1:100] 99.2 98.6 99.2 100.9 97.4 ...
## $ : num [1:100] 99.7 99.8 100.9 100.5 99.9 ...
## $ : num [1:100] 99.5 100.9 98.5 99.7 100 ...
- Como você pode criar um vetor indicando se cada coluna em um
data.frameé um fator?
# Flights por exemplo
flights %>%
# Lembrem-se que a pergunta pedia para criar um vetor!
map_lgl(is.factor)
## year month day dep_time sched_dep_time
## FALSE FALSE FALSE FALSE FALSE
## dep_delay arr_time sched_arr_time arr_delay carrier
## FALSE FALSE FALSE FALSE FALSE
## flight tailnum origin dest air_time
## FALSE FALSE FALSE FALSE FALSE
## distance hour minute time_hour
## FALSE FALSE FALSE FALSE
-
Usando as funções predicado
keepediscard:- Selecione todas as colunas caractere no banco
flights.
- Selecione todas as colunas caractere no banco
flights %>% keep(is.character)
## # A tibble: 336,776 x 4
## carrier tailnum origin dest
## <chr> <chr> <chr> <chr>
## 1 UA N14228 EWR IAH
## 2 UA N24211 LGA IAH
## 3 AA N619AA JFK MIA
## 4 B6 N804JB JFK BQN
## 5 DL N668DN LGA ATL
## 6 UA N39463 EWR ORD
## 7 B6 N516JB EWR FLL
## 8 EV N829AS LGA IAD
## 9 B6 N593JB JFK MCO
## 10 AA N3ALAA LGA ORD
## # ... with 336,766 more rows
2. Descarte os caracteres em `mpg`.
mpg %>% discard(is.character)
## # A tibble: 234 x 5
## displ year cyl cty hwy
## <dbl> <int> <int> <int> <int>
## 1 1.8 1999 4 18 29
## 2 1.8 1999 4 21 29
## 3 2 2008 4 20 31
## 4 2 2008 4 21 30
## 5 2.8 1999 6 16 26
## 6 2.8 1999 6 18 26
## 7 3.1 2008 6 18 27
## 8 1.8 1999 4 18 26
## 9 1.8 1999 4 16 25
## 10 2 2008 4 20 28
## # ... with 224 more rows
3. Selecione os fatores ordenados em `diamonds`.
diamonds %>% keep(is.ordered)
## # A tibble: 53,940 x 3
## cut color clarity
## <ord> <ord> <ord>
## 1 Ideal E SI2
## 2 Premium E SI1
## 3 Good E VS1
## 4 Premium I VS2
## 5 Good J SI2
## 6 Very Good J VVS2
## 7 Very Good I VVS1
## 8 Very Good H SI1
## 9 Fair E VS2
## 10 Very Good H VS1
## # ... with 53,930 more rows
4. Descarte as variáveis não-numéricas em `iris`
iris %>% discard(negate(is.numeric))
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## 1 5.1 3.5 1.4 0.2
## 2 4.9 3.0 1.4 0.2
## 3 4.7 3.2 1.3 0.2
## 4 4.6 3.1 1.5 0.2
## 5 5.0 3.6 1.4 0.2
## 6 5.4 3.9 1.7 0.4
## 7 4.6 3.4 1.4 0.3
## 8 5.0 3.4 1.5 0.2
## 9 4.4 2.9 1.4 0.2
## 10 4.9 3.1 1.5 0.1
## 11 5.4 3.7 1.5 0.2
## 12 4.8 3.4 1.6 0.2
## 13 4.8 3.0 1.4 0.1
## 14 4.3 3.0 1.1 0.1
## 15 5.8 4.0 1.2 0.2
## 16 5.7 4.4 1.5 0.4
## 17 5.4 3.9 1.3 0.4
## 18 5.1 3.5 1.4 0.3
## 19 5.7 3.8 1.7 0.3
## 20 5.1 3.8 1.5 0.3
## 21 5.4 3.4 1.7 0.2
## 22 5.1 3.7 1.5 0.4
## 23 4.6 3.6 1.0 0.2
## 24 5.1 3.3 1.7 0.5
## 25 4.8 3.4 1.9 0.2
## 26 5.0 3.0 1.6 0.2
## 27 5.0 3.4 1.6 0.4
## 28 5.2 3.5 1.5 0.2
## 29 5.2 3.4 1.4 0.2
## 30 4.7 3.2 1.6 0.2
## 31 4.8 3.1 1.6 0.2
## 32 5.4 3.4 1.5 0.4
## 33 5.2 4.1 1.5 0.1
## 34 5.5 4.2 1.4 0.2
## 35 4.9 3.1 1.5 0.2
## 36 5.0 3.2 1.2 0.2
## 37 5.5 3.5 1.3 0.2
## 38 4.9 3.6 1.4 0.1
## 39 4.4 3.0 1.3 0.2
## 40 5.1 3.4 1.5 0.2
## 41 5.0 3.5 1.3 0.3
## 42 4.5 2.3 1.3 0.3
## 43 4.4 3.2 1.3 0.2
## 44 5.0 3.5 1.6 0.6
## 45 5.1 3.8 1.9 0.4
## 46 4.8 3.0 1.4 0.3
## 47 5.1 3.8 1.6 0.2
## 48 4.6 3.2 1.4 0.2
## 49 5.3 3.7 1.5 0.2
## 50 5.0 3.3 1.4 0.2
## 51 7.0 3.2 4.7 1.4
## 52 6.4 3.2 4.5 1.5
## 53 6.9 3.1 4.9 1.5
## 54 5.5 2.3 4.0 1.3
## 55 6.5 2.8 4.6 1.5
## 56 5.7 2.8 4.5 1.3
## 57 6.3 3.3 4.7 1.6
## 58 4.9 2.4 3.3 1.0
## 59 6.6 2.9 4.6 1.3
## 60 5.2 2.7 3.9 1.4
## 61 5.0 2.0 3.5 1.0
## 62 5.9 3.0 4.2 1.5
## 63 6.0 2.2 4.0 1.0
## 64 6.1 2.9 4.7 1.4
## 65 5.6 2.9 3.6 1.3
## 66 6.7 3.1 4.4 1.4
## 67 5.6 3.0 4.5 1.5
## 68 5.8 2.7 4.1 1.0
## 69 6.2 2.2 4.5 1.5
## 70 5.6 2.5 3.9 1.1
## 71 5.9 3.2 4.8 1.8
## 72 6.1 2.8 4.0 1.3
## 73 6.3 2.5 4.9 1.5
## 74 6.1 2.8 4.7 1.2
## 75 6.4 2.9 4.3 1.3
## 76 6.6 3.0 4.4 1.4
## 77 6.8 2.8 4.8 1.4
## 78 6.7 3.0 5.0 1.7
## 79 6.0 2.9 4.5 1.5
## 80 5.7 2.6 3.5 1.0
## 81 5.5 2.4 3.8 1.1
## 82 5.5 2.4 3.7 1.0
## 83 5.8 2.7 3.9 1.2
## 84 6.0 2.7 5.1 1.6
## 85 5.4 3.0 4.5 1.5
## 86 6.0 3.4 4.5 1.6
## 87 6.7 3.1 4.7 1.5
## 88 6.3 2.3 4.4 1.3
## 89 5.6 3.0 4.1 1.3
## 90 5.5 2.5 4.0 1.3
## 91 5.5 2.6 4.4 1.2
## 92 6.1 3.0 4.6 1.4
## 93 5.8 2.6 4.0 1.2
## 94 5.0 2.3 3.3 1.0
## 95 5.6 2.7 4.2 1.3
## 96 5.7 3.0 4.2 1.2
## 97 5.7 2.9 4.2 1.3
## 98 6.2 2.9 4.3 1.3
## 99 5.1 2.5 3.0 1.1
## 100 5.7 2.8 4.1 1.3
## 101 6.3 3.3 6.0 2.5
## 102 5.8 2.7 5.1 1.9
## 103 7.1 3.0 5.9 2.1
## 104 6.3 2.9 5.6 1.8
## 105 6.5 3.0 5.8 2.2
## 106 7.6 3.0 6.6 2.1
## 107 4.9 2.5 4.5 1.7
## 108 7.3 2.9 6.3 1.8
## 109 6.7 2.5 5.8 1.8
## 110 7.2 3.6 6.1 2.5
## 111 6.5 3.2 5.1 2.0
## 112 6.4 2.7 5.3 1.9
## 113 6.8 3.0 5.5 2.1
## 114 5.7 2.5 5.0 2.0
## 115 5.8 2.8 5.1 2.4
## 116 6.4 3.2 5.3 2.3
## 117 6.5 3.0 5.5 1.8
## 118 7.7 3.8 6.7 2.2
## 119 7.7 2.6 6.9 2.3
## 120 6.0 2.2 5.0 1.5
## 121 6.9 3.2 5.7 2.3
## 122 5.6 2.8 4.9 2.0
## 123 7.7 2.8 6.7 2.0
## 124 6.3 2.7 4.9 1.8
## 125 6.7 3.3 5.7 2.1
## 126 7.2 3.2 6.0 1.8
## 127 6.2 2.8 4.8 1.8
## 128 6.1 3.0 4.9 1.8
## 129 6.4 2.8 5.6 2.1
## 130 7.2 3.0 5.8 1.6
## 131 7.4 2.8 6.1 1.9
## 132 7.9 3.8 6.4 2.0
## 133 6.4 2.8 5.6 2.2
## 134 6.3 2.8 5.1 1.5
## 135 6.1 2.6 5.6 1.4
## 136 7.7 3.0 6.1 2.3
## 137 6.3 3.4 5.6 2.4
## 138 6.4 3.1 5.5 1.8
## 139 6.0 3.0 4.8 1.8
## 140 6.9 3.1 5.4 2.1
## 141 6.7 3.1 5.6 2.4
## 142 6.9 3.1 5.1 2.3
## 143 5.8 2.7 5.1 1.9
## 144 6.8 3.2 5.9 2.3
## 145 6.7 3.3 5.7 2.5
## 146 6.7 3.0 5.2 2.3
## 147 6.3 2.5 5.0 1.9
## 148 6.5 3.0 5.2 2.0
## 149 6.2 3.4 5.4 2.3
## 150 5.9 3.0 5.1 1.8
- Imagine que você tem um diretório cheio de arquivos
.csvque correspondem a um único banco de dados. Você tem os caminhos de todos eles num vetor com a formac(arquivo_1.csv, ..., arquivo_n.csv). Como você importaria esses arquivos? Tente fazer duas soluções diferentes.
# Vamos usar um diretório temporário que vamos preencher com vários tibbles
pasta <- tempdir()
# Criamos 100 tibbles e guardamos em 100 arquivos csv.
x <- rep(10, 100)
arqs <- sprintf("arquivo%s.csv", 1:100)
map(x, ~tibble(var1 = rnorm(.x), var2 = rnorm(.x))) %>%
map2(arqs, ~write_csv(.x, file.path(pasta, .y))) %>%
str()
# Nossos arquivos estão salvos no diretório temporário:
dir(pasta)
## [1] "arquivo1.csv" "arquivo10.csv" "arquivo100.csv" "arquivo11.csv"
## [5] "arquivo12.csv" "arquivo13.csv" "arquivo14.csv" "arquivo15.csv"
## [9] "arquivo16.csv" "arquivo17.csv" "arquivo18.csv" "arquivo19.csv"
## [13] "arquivo2.csv" "arquivo20.csv" "arquivo21.csv" "arquivo22.csv"
## [17] "arquivo23.csv" "arquivo24.csv" "arquivo25.csv" "arquivo26.csv"
## [21] "arquivo27.csv" "arquivo28.csv" "arquivo29.csv" "arquivo3.csv"
## [25] "arquivo30.csv" "arquivo31.csv" "arquivo32.csv" "arquivo33.csv"
## [29] "arquivo34.csv" "arquivo35.csv" "arquivo36.csv" "arquivo37.csv"
## [33] "arquivo38.csv" "arquivo39.csv" "arquivo4.csv" "arquivo40.csv"
## [37] "arquivo41.csv" "arquivo42.csv" "arquivo43.csv" "arquivo44.csv"
## [41] "arquivo45.csv" "arquivo46.csv" "arquivo47.csv" "arquivo48.csv"
## [45] "arquivo49.csv" "arquivo5.csv" "arquivo50.csv" "arquivo51.csv"
## [49] "arquivo52.csv" "arquivo53.csv" "arquivo54.csv" "arquivo55.csv"
## [53] "arquivo56.csv" "arquivo57.csv" "arquivo58.csv" "arquivo59.csv"
## [57] "arquivo6.csv" "arquivo60.csv" "arquivo61.csv" "arquivo62.csv"
## [61] "arquivo63.csv" "arquivo64.csv" "arquivo65.csv" "arquivo66.csv"
## [65] "arquivo67.csv" "arquivo68.csv" "arquivo69.csv" "arquivo7.csv"
## [69] "arquivo70.csv" "arquivo71.csv" "arquivo72.csv" "arquivo73.csv"
## [73] "arquivo74.csv" "arquivo75.csv" "arquivo76.csv" "arquivo77.csv"
## [77] "arquivo78.csv" "arquivo79.csv" "arquivo8.csv" "arquivo80.csv"
## [81] "arquivo81.csv" "arquivo82.csv" "arquivo83.csv" "arquivo84.csv"
## [85] "arquivo85.csv" "arquivo86.csv" "arquivo87.csv" "arquivo88.csv"
## [89] "arquivo89.csv" "arquivo9.csv" "arquivo90.csv" "arquivo91.csv"
## [93] "arquivo92.csv" "arquivo93.csv" "arquivo94.csv" "arquivo95.csv"
## [97] "arquivo96.csv" "arquivo97.csv" "arquivo98.csv" "arquivo99.csv"
## [101] "filecf810bd36a5" "filecf81149221d" "filecf81164e23" "filecf8161f65f7"
## [105] "filecf8171030fd" "filecf819e43f49" "filecf81ad26a9f" "filecf81bed9f1"
## [109] "filecf81e7a7e68" "filecf81ef03542" "filecf81fa64a3e" "filecf8239d75ab"
## [113] "filecf8261936" "filecf826d55378" "filecf8291061a0" "filecf82b985019"
## [117] "filecf82c00737b" "filecf8339f5" "filecf8341e67d6" "filecf8344158ac"
## [121] "filecf837a47cc7" "filecf837f15510" "filecf8387a3d29" "filecf83a71232b"
## [125] "filecf83ac3253a" "filecf83af94598" "filecf83f794f64" "filecf8402156c1"
## [129] "filecf840c15566" "filecf84163761c" "filecf841d3b53" "filecf844d5642"
## [133] "filecf845e73bac" "filecf8462819d6" "filecf84b8b3144" "filecf84ddc73a0"
## [137] "filecf84fe515d4" "filecf8500a5a10" "filecf8512766ea" "filecf852fe119b"
## [141] "filecf8550a39d" "filecf85b14fec" "filecf85e053c83" "filecf85e6a12f4"
## [145] "filecf8639c6053" "filecf863dc5f3b" "filecf8646e477f" "filecf865645c5d"
## [149] "filecf868634c0d" "filecf869137c8a" "filecf86a7852d4" "filecf86b5b250"
## [153] "filecf86b9e3bb7" "filecf86d216ac" "filecf86e894a16" "filecf86ea916d3"
## [157] "filecf86f22266" "filecf86f975377" "filecf8757a72be" "filecf876956e82"
## [161] "filecf876967b1" "filecf8787a1370" "filecf879037c39" "filecf87964711"
## [165] "filecf87afe7046" "filecf88456ae0" "filecf898344ab"
# Agora vamos ler todos de volta para uma tibble só.
arqs <- dir(pasta, pattern = ".csv", full.names = T)
# Juntando linhas
df <- map_dfr(arqs, read_csv)
df
## # A tibble: 1,000 x 2
## var1 var2
## <dbl> <dbl>
## 1 0.837 -0.554
## 2 -1.12 1.08
## 3 -0.821 1.21
## 4 0.384 0.465
## 5 0.885 0.477
## 6 -0.194 0.517
## 7 0.710 0.128
## 8 -1.04 -0.947
## 9 0.782 1.58
## 10 0.571 0.990
## # ... with 990 more rows
# Juntando colunas
df <- map_dfc(arqs, read_csv)
df
## # A tibble: 10 x 200
## var1...1 var2...2 var1...3 var2...4 var1...5 var2...6 var1...7 var2...8
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 0.837 -0.554 -1.35 0.522 1.49 0.648 -0.567 -0.868
## 2 -1.12 1.08 0.876 2.11 0.0449 0.0737 -0.808 0.177
## 3 -0.821 1.21 1.77 -1.31 1.51 1.35 0.559 -0.119
## 4 0.384 0.465 -0.496 1.10 1.13 -0.331 0.427 0.131
## 5 0.885 0.477 -0.514 -0.573 -0.0704 0.581 0.751 0.629
## 6 -0.194 0.517 0.362 -1.62 -0.725 -2.37 0.710 -0.293
## 7 0.710 0.128 1.11 0.144 0.00462 1.66 1.32 -0.414
## 8 -1.04 -0.947 0.00775 -1.27 0.0365 0.0813 -1.10 0.757
## 9 0.782 1.58 0.695 -0.375 -0.342 0.341 -0.325 0.834
## 10 0.571 0.990 -0.475 -0.356 -1.17 -0.827 -0.148 -1.29
## # ... with 192 more variables: var1...9 <dbl>, var2...10 <dbl>,
## # var1...11 <dbl>, var2...12 <dbl>, var1...13 <dbl>, var2...14 <dbl>,
## # var1...15 <dbl>, var2...16 <dbl>, var1...17 <dbl>, var2...18 <dbl>,
## # var1...19 <dbl>, var2...20 <dbl>, var1...21 <dbl>, var2...22 <dbl>,
## # var1...23 <dbl>, var2...24 <dbl>, var1...25 <dbl>, var2...26 <dbl>,
## # var1...27 <dbl>, var2...28 <dbl>, var1...29 <dbl>, var2...30 <dbl>,
## # var1...31 <dbl>, var2...32 <dbl>, var1...33 <dbl>, var2...34 <dbl>,
## # var1...35 <dbl>, var2...36 <dbl>, var1...37 <dbl>, var2...38 <dbl>,
## # var1...39 <dbl>, var2...40 <dbl>, var1...41 <dbl>, var2...42 <dbl>,
## # var1...43 <dbl>, var2...44 <dbl>, var1...45 <dbl>, var2...46 <dbl>,
## # var1...47 <dbl>, var2...48 <dbl>, var1...49 <dbl>, var2...50 <dbl>,
## # var1...51 <dbl>, var2...52 <dbl>, var1...53 <dbl>, var2...54 <dbl>,
## # var1...55 <dbl>, var2...56 <dbl>, var1...57 <dbl>, var2...58 <dbl>,
## # var1...59 <dbl>, var2...60 <dbl>, var1...61 <dbl>, var2...62 <dbl>,
## # var1...63 <dbl>, var2...64 <dbl>, var1...65 <dbl>, var2...66 <dbl>,
## # var1...67 <dbl>, var2...68 <dbl>, var1...69 <dbl>, var2...70 <dbl>,
## # var1...71 <dbl>, var2...72 <dbl>, var1...73 <dbl>, var2...74 <dbl>,
## # var1...75 <dbl>, var2...76 <dbl>, var1...77 <dbl>, var2...78 <dbl>,
## # var1...79 <dbl>, var2...80 <dbl>, var1...81 <dbl>, var2...82 <dbl>,
## # var1...83 <dbl>, var2...84 <dbl>, var1...85 <dbl>, var2...86 <dbl>,
## # var1...87 <dbl>, var2...88 <dbl>, var1...89 <dbl>, var2...90 <dbl>,
## # var1...91 <dbl>, var2...92 <dbl>, var1...93 <dbl>, var2...94 <dbl>,
## # var1...95 <dbl>, var2...96 <dbl>, var1...97 <dbl>, var2...98 <dbl>,
## # var1...99 <dbl>, var2...100 <dbl>, var1...101 <dbl>, var2...102 <dbl>,
## # var1...103 <dbl>, var2...104 <dbl>, var1...105 <dbl>, var2...106 <dbl>,
## # var1...107 <dbl>, var2...108 <dbl>, ...
# Usando reduce
# linhas
df <- map(arqs, read_csv) %>% reduce(bind_rows)
df
## # A tibble: 1,000 x 2
## var1 var2
## <dbl> <dbl>
## 1 0.837 -0.554
## 2 -1.12 1.08
## 3 -0.821 1.21
## 4 0.384 0.465
## 5 0.885 0.477
## 6 -0.194 0.517
## 7 0.710 0.128
## 8 -1.04 -0.947
## 9 0.782 1.58
## 10 0.571 0.990
## # ... with 990 more rows
# colunas
df <- map(arqs, read_csv) %>% reduce(bind_cols)
df
## # A tibble: 10 x 200
## var1...1 var2...2 var1...3 var2...4 var1...5 var2...6 var1...7 var2...8
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 0.837 -0.554 -1.35 0.522 1.49 0.648 -0.567 -0.868
## 2 -1.12 1.08 0.876 2.11 0.0449 0.0737 -0.808 0.177
## 3 -0.821 1.21 1.77 -1.31 1.51 1.35 0.559 -0.119
## 4 0.384 0.465 -0.496 1.10 1.13 -0.331 0.427 0.131
## 5 0.885 0.477 -0.514 -0.573 -0.0704 0.581 0.751 0.629
## 6 -0.194 0.517 0.362 -1.62 -0.725 -2.37 0.710 -0.293
## 7 0.710 0.128 1.11 0.144 0.00462 1.66 1.32 -0.414
## 8 -1.04 -0.947 0.00775 -1.27 0.0365 0.0813 -1.10 0.757
## 9 0.782 1.58 0.695 -0.375 -0.342 0.341 -0.325 0.834
## 10 0.571 0.990 -0.475 -0.356 -1.17 -0.827 -0.148 -1.29
## # ... with 192 more variables: var1...9 <dbl>, var2...10 <dbl>,
## # var1...11 <dbl>, var2...12 <dbl>, var1...13 <dbl>, var2...14 <dbl>,
## # var1...15 <dbl>, var2...16 <dbl>, var1...17 <dbl>, var2...18 <dbl>,
## # var1...19 <dbl>, var2...20 <dbl>, var1...21 <dbl>, var2...22 <dbl>,
## # var1...23 <dbl>, var2...24 <dbl>, var1...25 <dbl>, var2...26 <dbl>,
## # var1...27 <dbl>, var2...28 <dbl>, var1...29 <dbl>, var2...30 <dbl>,
## # var1...31 <dbl>, var2...32 <dbl>, var1...33 <dbl>, var2...34 <dbl>,
## # var1...35 <dbl>, var2...36 <dbl>, var1...37 <dbl>, var2...38 <dbl>,
## # var1...39 <dbl>, var2...40 <dbl>, var1...41 <dbl>, var2...42 <dbl>,
## # var1...43 <dbl>, var2...44 <dbl>, var1...45 <dbl>, var2...46 <dbl>,
## # var1...47 <dbl>, var2...48 <dbl>, var1...49 <dbl>, var2...50 <dbl>,
## # var1...51 <dbl>, var2...52 <dbl>, var1...53 <dbl>, var2...54 <dbl>,
## # var1...55 <dbl>, var2...56 <dbl>, var1...57 <dbl>, var2...58 <dbl>,
## # var1...59 <dbl>, var2...60 <dbl>, var1...61 <dbl>, var2...62 <dbl>,
## # var1...63 <dbl>, var2...64 <dbl>, var1...65 <dbl>, var2...66 <dbl>,
## # var1...67 <dbl>, var2...68 <dbl>, var1...69 <dbl>, var2...70 <dbl>,
## # var1...71 <dbl>, var2...72 <dbl>, var1...73 <dbl>, var2...74 <dbl>,
## # var1...75 <dbl>, var2...76 <dbl>, var1...77 <dbl>, var2...78 <dbl>,
## # var1...79 <dbl>, var2...80 <dbl>, var1...81 <dbl>, var2...82 <dbl>,
## # var1...83 <dbl>, var2...84 <dbl>, var1...85 <dbl>, var2...86 <dbl>,
## # var1...87 <dbl>, var2...88 <dbl>, var1...89 <dbl>, var2...90 <dbl>,
## # var1...91 <dbl>, var2...92 <dbl>, var1...93 <dbl>, var2...94 <dbl>,
## # var1...95 <dbl>, var2...96 <dbl>, var1...97 <dbl>, var2...98 <dbl>,
## # var1...99 <dbl>, var2...100 <dbl>, var1...101 <dbl>, var2...102 <dbl>,
## # var1...103 <dbl>, var2...104 <dbl>, var1...105 <dbl>, var2...106 <dbl>,
## # var1...107 <dbl>, var2...108 <dbl>, ...
- Escreva um código sucinto que implemente vários modelos lineares especificados por você. Salve os resultados numa
tibblecom colunas-lista. Depois, extraia os resultados comunnest(). Use o exemplo como guia.
mtcars
## mpg cyl disp hp drat wt qsec vs am gear carb
## Mazda RX4 21.0 6 160.0 110 3.90 2.620 16.46 0 1 4 4
## Mazda RX4 Wag 21.0 6 160.0 110 3.90 2.875 17.02 0 1 4 4
## Datsun 710 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1
## Hornet 4 Drive 21.4 6 258.0 110 3.08 3.215 19.44 1 0 3 1
## Hornet Sportabout 18.7 8 360.0 175 3.15 3.440 17.02 0 0 3 2
## Valiant 18.1 6 225.0 105 2.76 3.460 20.22 1 0 3 1
## Duster 360 14.3 8 360.0 245 3.21 3.570 15.84 0 0 3 4
## Merc 240D 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2
## Merc 230 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2
## Merc 280 19.2 6 167.6 123 3.92 3.440 18.30 1 0 4 4
## Merc 280C 17.8 6 167.6 123 3.92 3.440 18.90 1 0 4 4
## Merc 450SE 16.4 8 275.8 180 3.07 4.070 17.40 0 0 3 3
## Merc 450SL 17.3 8 275.8 180 3.07 3.730 17.60 0 0 3 3
## Merc 450SLC 15.2 8 275.8 180 3.07 3.780 18.00 0 0 3 3
## Cadillac Fleetwood 10.4 8 472.0 205 2.93 5.250 17.98 0 0 3 4
## Lincoln Continental 10.4 8 460.0 215 3.00 5.424 17.82 0 0 3 4
## Chrysler Imperial 14.7 8 440.0 230 3.23 5.345 17.42 0 0 3 4
## Fiat 128 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1
## Honda Civic 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2
## Toyota Corolla 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1
## Toyota Corona 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1
## Dodge Challenger 15.5 8 318.0 150 2.76 3.520 16.87 0 0 3 2
## AMC Javelin 15.2 8 304.0 150 3.15 3.435 17.30 0 0 3 2
## Camaro Z28 13.3 8 350.0 245 3.73 3.840 15.41 0 0 3 4
## Pontiac Firebird 19.2 8 400.0 175 3.08 3.845 17.05 0 0 3 2
## Fiat X1-9 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1
## Porsche 914-2 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2
## Lotus Europa 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2
## Ford Pantera L 15.8 8 351.0 264 4.22 3.170 14.50 0 1 5 4
## Ferrari Dino 19.7 6 145.0 175 3.62 2.770 15.50 0 1 5 6
## Maserati Bora 15.0 8 301.0 335 3.54 3.570 14.60 0 1 5 8
## Volvo 142E 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2
modelos <- c("mpg ~ wt",
"mpg ~ wt + cyl",
"mpg ~ wt + cyl + drat",
"mpg ~ wt + cyl + drat + am")
# Estou usando funções do pacote broom, mas você não era obrigado a apresentar
# os resultados bonitinho. O objetivo era treinar esse tipo de workflow.
modelos_saida <- tibble(
especificacao = modelos,
ajuste = map(especificacao, lm, data = mtcars),
coefs = map(ajuste, broom::tidy),
stats = map(ajuste, broom::glance),
aums = map(ajuste, broom::augment)
)
modelos_saida %>%
select(especificacao, coefs) %>%
unnest(coefs) %>%
filter(term != "(Intercept)")
## # A tibble: 10 x 6
## especificacao term estimate std.error statistic p.value
## <chr> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 mpg ~ wt wt -5.34 0.559 -9.56 1.29e-10
## 2 mpg ~ wt + cyl wt -3.19 0.757 -4.22 2.22e- 4
## 3 mpg ~ wt + cyl cyl -1.51 0.415 -3.64 1.06e- 3
## 4 mpg ~ wt + cyl + drat wt -3.19 0.829 -3.85 6.24e- 4
## 5 mpg ~ wt + cyl + drat cyl -1.51 0.446 -3.38 2.14e- 3
## 6 mpg ~ wt + cyl + drat drat -0.0162 1.32 -0.0122 9.90e- 1
## 7 mpg ~ wt + cyl + drat + am wt -3.13 0.932 -3.36 2.32e- 3
## 8 mpg ~ wt + cyl + drat + am cyl -1.53 0.465 -3.28 2.88e- 3
## 9 mpg ~ wt + cyl + drat + am drat -0.130 1.53 -0.0851 9.33e- 1
## 10 mpg ~ wt + cyl + drat + am am 0.237 1.51 0.157 8.76e- 1
modelos_saida %>%
select(especificacao, stats) %>%
unnest(stats)
## # A tibble: 4 x 13
## especificacao r.squared adj.r.squared sigma statistic p.value df logLik
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 mpg ~ wt 0.753 0.745 3.05 91.4 1.29e-10 1 -80.0
## 2 mpg ~ wt + cyl 0.830 0.819 2.57 70.9 6.81e-12 2 -74.0
## 3 mpg ~ wt + cyl ~ 0.830 0.812 2.61 45.6 6.57e-11 3 -74.0
## 4 mpg ~ wt + cyl ~ 0.830 0.805 2.66 33.0 4.84e-10 4 -74.0
## # ... with 5 more variables: AIC <dbl>, BIC <dbl>, deviance <dbl>,
## # df.residual <int>, nobs <int>
modelos_saida %>%
select(especificacao, aums) %>%
filter(especificacao == "mpg ~ wt + cyl + drat + am") %>%
unnest(aums)
## # A tibble: 32 x 13
## especificacao .rownames mpg wt cyl drat am .fitted .resid .hat
## <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 mpg ~ wt + cyl~ Mazda RX4 21 2.62 6 3.9 1 22.3 -1.35 0.0899
## 2 mpg ~ wt + cyl~ Mazda RX~ 21 2.88 6 3.9 1 21.6 -0.550 0.0924
## 3 mpg ~ wt + cyl~ Datsun 7~ 22.8 2.32 4 3.85 1 26.3 -3.55 0.137
## 4 mpg ~ wt + cyl~ Hornet 4~ 21.4 3.22 6 3.08 0 20.4 1.05 0.113
## 5 mpg ~ wt + cyl~ Hornet S~ 18.7 3.44 8 3.15 0 16.6 2.11 0.122
## 6 mpg ~ wt + cyl~ Valiant 18.1 3.46 6 2.76 0 19.6 -1.53 0.207
## 7 mpg ~ wt + cyl~ Duster 3~ 14.3 3.57 8 3.21 0 16.2 -1.87 0.103
## 8 mpg ~ wt + cyl~ Merc 240D 24.4 3.19 4 3.69 0 23.4 0.996 0.189
## 9 mpg ~ wt + cyl~ Merc 230 22.8 3.15 4 3.92 0 23.5 -0.700 0.207
## 10 mpg ~ wt + cyl~ Merc 280 19.2 3.44 6 3.92 0 19.5 -0.340 0.149
## # ... with 22 more rows, and 3 more variables: .sigma <dbl>, .cooksd <dbl>,
## # .std.resid <dbl>